(邦訳:立体構造情報に基づくタンパク質間相互作用ネットワーク予測)
| 大上 雅史 日本学術振興会 特別研究員(PD)/東京工業大学 特別研究員 |
[背景]タンパク質間相互作用(PPI)の網羅的解明が生命科学の重要課題
[問題]大量の構造情報を扱えるバイオインフォマティクスツールの開発
[貢献]PPI予測計算の高速化と大規模並列実装,および実問題への応用
生体内のタンパク質間の相互作用(Protein-Protein Interaction, PPI)の網羅的な解明は,生物学や医学・薬学における重要課題である.徐々に解明されつつあるPPI情報は,がんや自己免疫疾患をはじめとするPPIの変調が原因とされる疾病に対して,病因の解明や新たな薬剤標的の探索を進める上でのきわめて貴重な情報源となっている.これらのPPIを計算機によって予測するにはタンパク質の立体構造情報の利用が必要不可欠だが,立体構造を精密に扱う従来の分子シミュレーション手法を単に利用するだけでは,1組のPPIの予測にすら数時間から数日を要し,網羅的なPPI予測を行うことは全く非現実的だと思われていた.
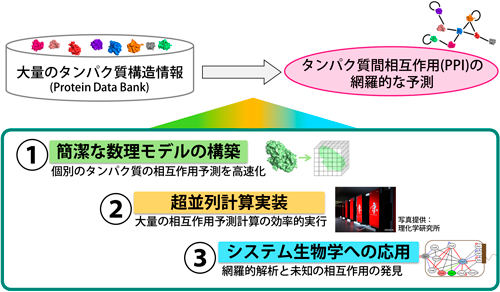
本研究は,高速なPPI予測手法を開発してヒト等の細胞内のすべてのタンパク質ペア間における PPIの網羅的な予測を実現することを目的とした.まずPPI予測を高速に行うための数理モデルを提案し,次に近年急速に発展するスーパーコンピュータ「京」や東工大TSUBAME等の超並列計算機上で効率良く動作させることで,この目的を達成した.
本研究の成果は,主に以下の3つの項目に分けられる.
本研究は,高速なPPI予測手法を開発してヒト等の細胞内のすべてのタンパク質ペア間における PPIの網羅的な予測を実現することを目的とした.まずPPI予測を高速に行うための数理モデルを提案し,次に近年急速に発展するスーパーコンピュータ「京」や東工大TSUBAME等の超並列計算機上で効率良く動作させることで,この目的を達成した.
本研究の成果は,主に以下の3つの項目に分けられる.
- タンパク質1ペアのPPIを高速に予測する新しい数理モデルの構築タンパク質構造を3次元格子上でモデル表現し,格子モデル上で形状の相補性を計算するrPSC(real Pairwise Shape Complementarity)モデルを新規に提案した.また,畳込み演算をフーリエ空間上で計算する技法を応用し,形状相補性を含む3つの物理化学的効果をたった1回の畳込み演算によって近似的に計算する方法を考案し,わずか数分でPPI予測を行うことに成功した.本研究で開発したPPI予測ソフトウェア「MEGADOCK」は,オープンソースで利用可能である.
- 超並列計算機への実装大量のPPIを予測するため,スーパーコンピュータ上で効率的に計算を実施する並列実装を行った.東工大TSUBAMEと「京」の両システムにおいて,ともに強スケーリング約90%以上という高い並列化効率を達成し,特に「京」では約66万CPUコアの同時利用という世界最大規模の計算を高効率に実現した.
- 細胞内PPIネットワーク予測への応用本研究で開発した予測技術を,実際のネットワーク解析に応用し,計算による未知PPIの発見を目指してきた.これまでに細菌の鞭毛モーターの働きに関するネットワーク(約1万ペア)や,ヒトの細胞死に関わるネットワーク(約4万ペア)に応用し,未知PPIの可能性が高いタンパク質ペアを発見してきた.また,肺がんに関わるネットワーク(約400万ペア)の解析も進行中であり,すでに11例のPPI候補を発見した.
本研究によって数百万件規模のPPI予測が初めて可能となった.本研究は,たとえばがん患者の細胞で発現しているタンパク質群を対象とした予測によって,がん細胞の制御機構の解明や,予測によって新規に発見されたPPIを標的とする抗がん剤創薬研究の新たな道が切り拓かれるなど,医学生物学分野のさまざまな問題を解決する可能性を持つ.
(2014年5月29日受付)

