大阪府の特定健康診査データの因果探索
1.因果探索の必要性
相関と因果は違うというのは,統計学を習った経験があるなら,誰しも知っていることであるが,相関をデータから取り出すのは簡単でも,因果をデータから取り出すのが可能なのか疑問に思われるかもしれない.実は,ある種の妥当な仮定の下で,データだけからかなりの因果的構造を推定することができる.
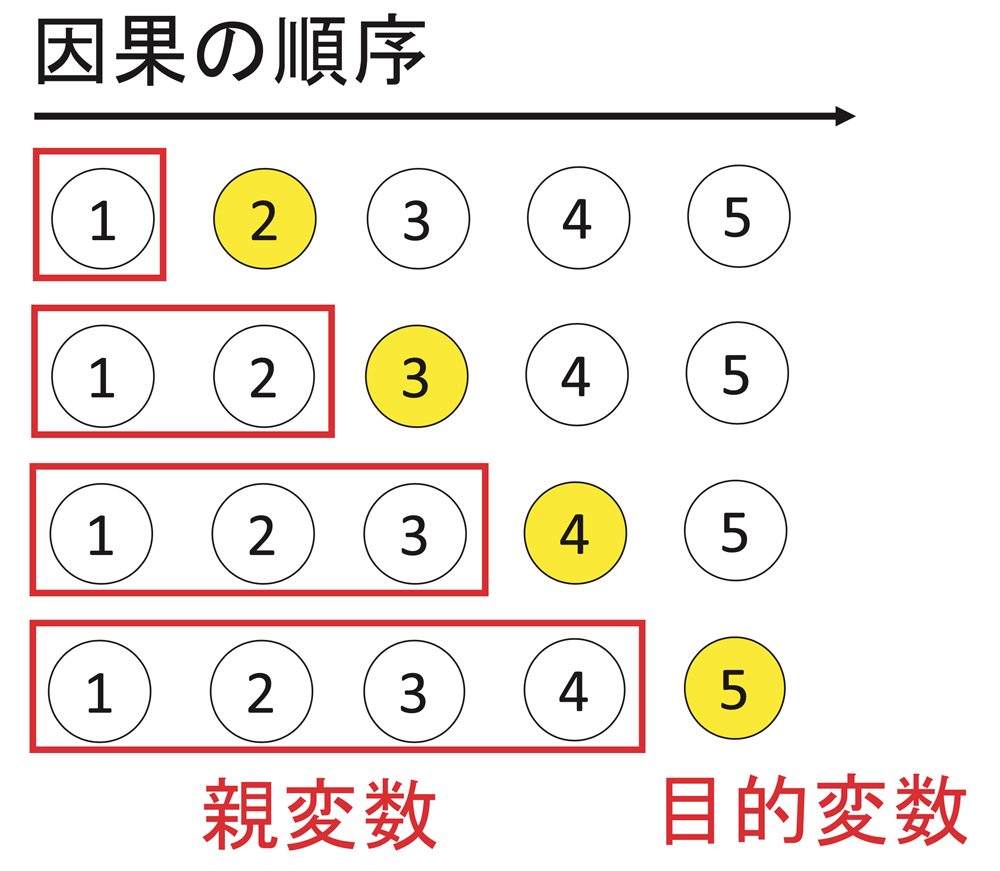
因果の構造を,原因となる変数と結果となる変数を矢印でつないで変数間の原因と結果の形を図形状に表したものを因果ダイアグラムと呼び,データから因果ダイアグラムの推定を行うことを因果探索と呼ぶ.厳密にはあまり区別されての使用はされていないが,傾向スコアのように解析者が因果モデルを仮定し,はたして因果があると言ってよいかどうか推測する作業のことを因果推論と呼ぶ.たとえば,医療ビッグデータのように非常に多くの要因が絡み合っているような問題を取り扱う場合には,あらかじめ因果関係を仮定することなしにデータ間の因果関係のネットワークを抽出する因果探索を実践できれば大変有用である.
本稿では,筆者らの行った大阪府全体の特定健康診査データ(以下,特定健診データ)への因果探索の応用[1]について実体験を交えながら赤裸々に語る.数値データのみから自動的に因果ダイアグラムを構築する因果探索のモデルとして使用したDirectLiNGAM[2],[3],[4],[5]の数理的側面について次章で簡単に紹介し,その後,陥りがちなピットフォールについて注意を喚起しつつ,解析の全容についてお伝えする.
2.LiNGAMの数理
2.1 LiNGAMの構造方程式
ここから,因果探索のモデルである LiNGAM の解説に入る.LiNGAM は,考えたい変数 間の因果関係として,式 (1) のような線形の関係を仮定するモデルである.このとき,外生変数と呼ばれる項 は,非ガウス分布に従うと仮定する.LiNGAM の構造方程式では,外生変数 が,いわば外から与える初期値で,これらはある確率分布からのサンプルとして実現される.我々が観測する量は,これらの線形結合として内生されるというのが,LiNGAM の世界観である.ここで, は,変数 から変数 への直接効果を表現する係数で,自分自身へのループのようなフィードバックは考えないことにする.これを式で表すと,
(1)
のように書ける.このままではプログラムする上で計算上の見通しが悪いので,内容は同じであるが,行列での表現に書き換えたものが式(2)または式(3)である.式(1)での
の部分は,行列
の対角成分が0として表現されている.
(2)
あるいは,
(3)ただし,
(4)
となる.
LiNGAMの目指すところは,外生変数 が非ガウス分布に従うという仮定のもとで,与えられた観測量 から,係数行列 を推定することである.
2.2 回帰による因果順序の推定
因果ダイアグラムの推定法にはさまざまな方法が考案されているが,今回の解析では,「2変数の因果関係を考えた場合,説明変数が原因である場合には残差と説明変数は独立だが,説明変数を結果にしてしまった場合,説明変数と残差が従属する」という性質を用いる.
どのようなことか,例を通して見てみよう. を独立な外生変数とし,次のように確率変数 を生成するLiNGAMを考える.
(5)
このモデルを,
として,
は[-0.5,0.5]の一様乱数からそれぞれ10,000個生成して,シミュレーションを行ったものが図1である.
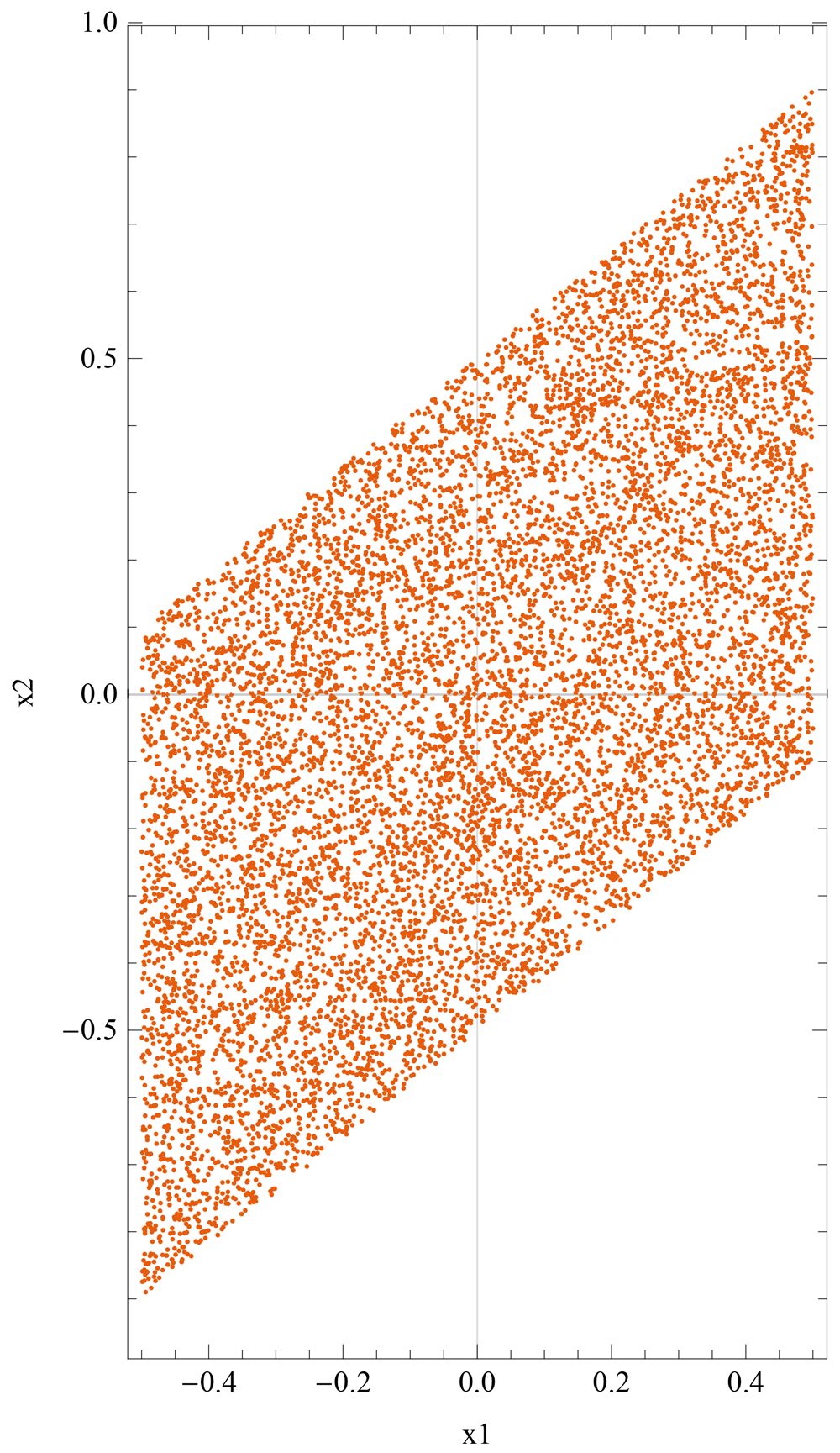
2.2.1 原因となる変数が説明変数のとき
このとき,回帰直線は の形になるから,残差は である.仮定から, は独立であるから, も独立になる.この様子を示したのが図2である.図下の散布図から分かるように, 軸,残差軸のどちらに平行な直線で切っても,同じ確率密度分布が得られるから,このとき,説明変数と残差は独立であることが見てとれる.
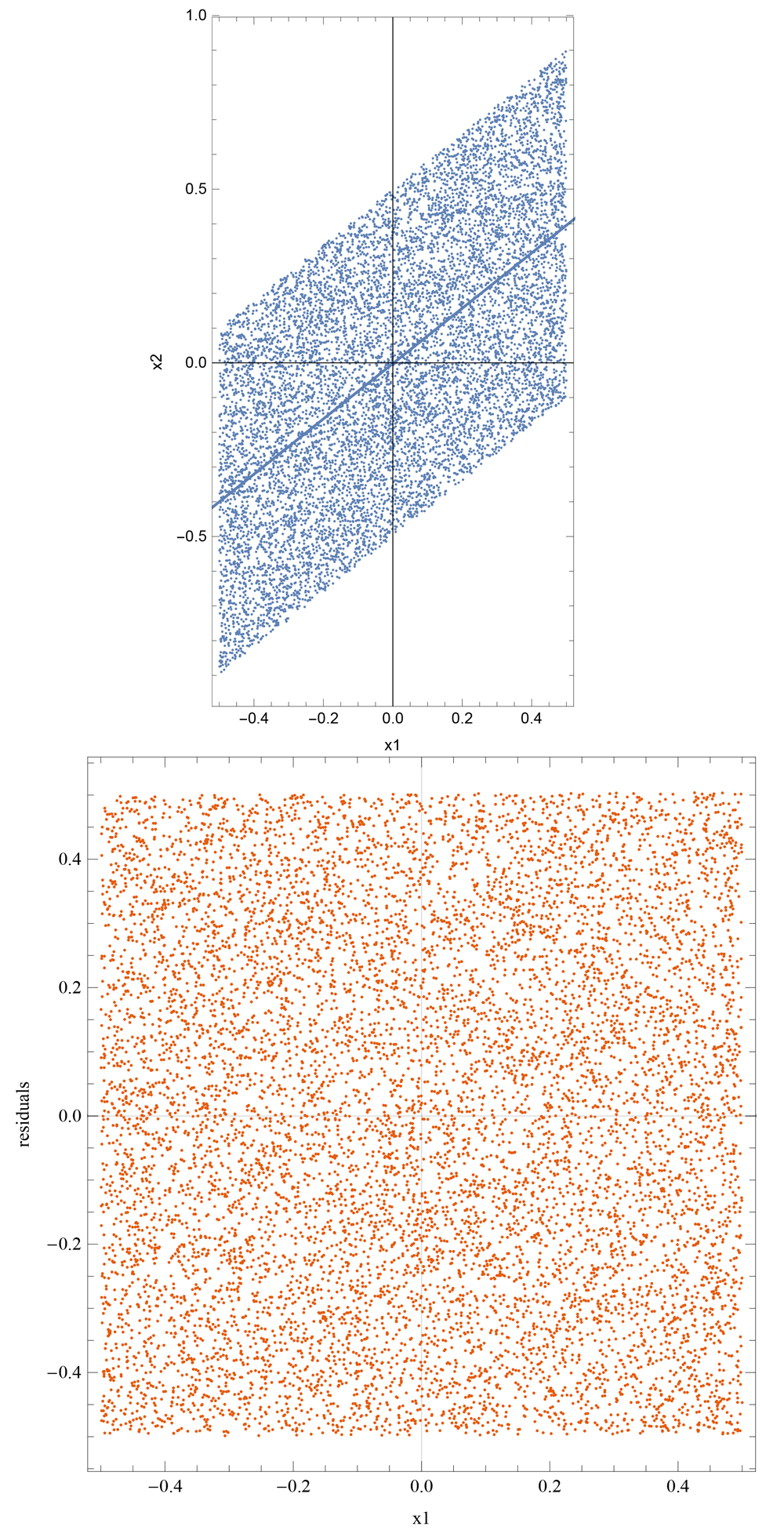
2.2.2 結果となる変数が説明変数のとき
次は, という形に回帰してしまったときのことを考えよう.
シミュレーションしたデータに対する回帰の例で示したのが,図3である.特に,下の図を見ると, 一定直線で切った切り口で,残差の確率密度分布が大きく変わってしまうことから,残差と説明変数が独立でないことが見てとれる.
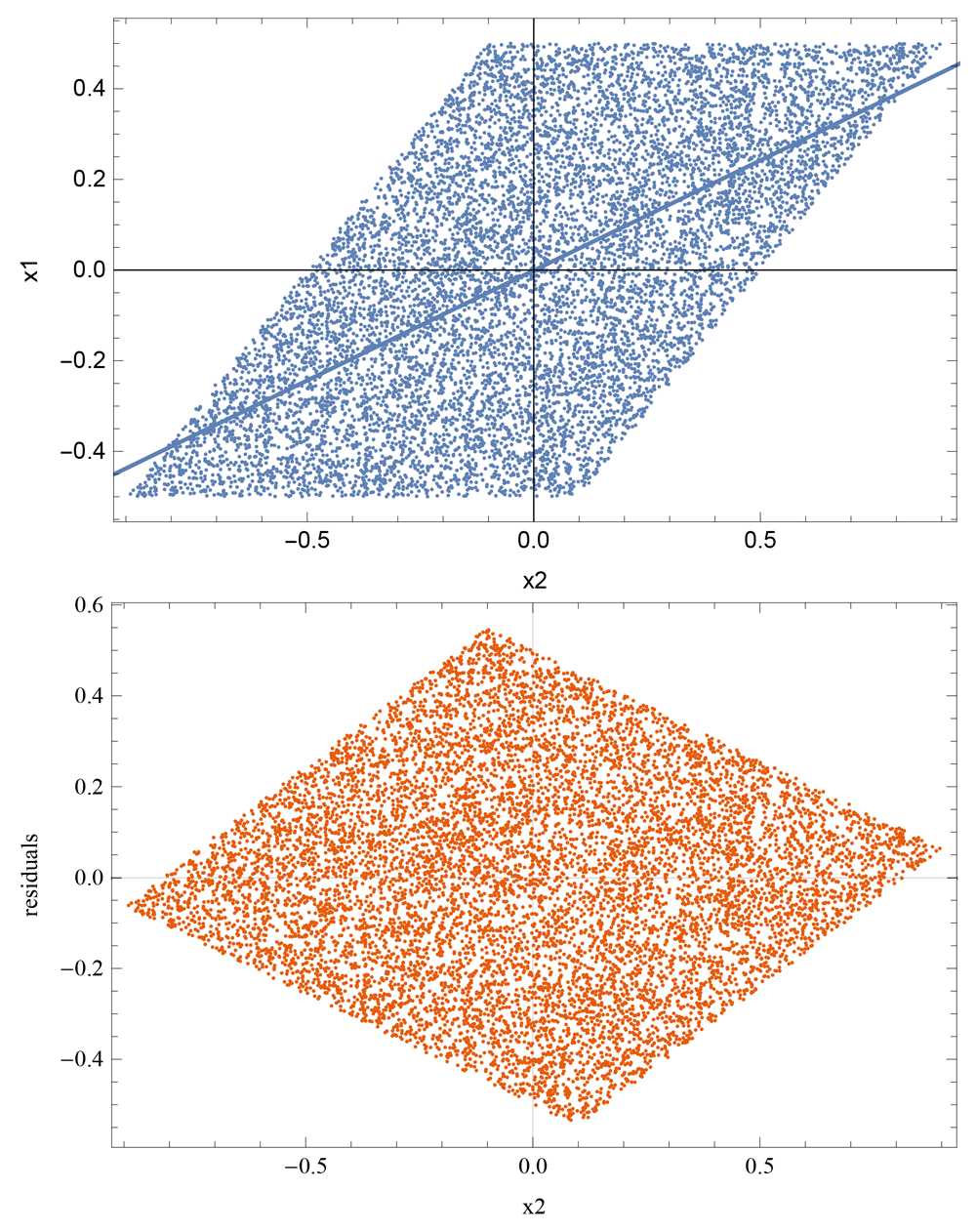
この話を一般化して,変数の数が3つ以上の場合にも,因果の最初の変数を推定することができる.
定理 LiNGAM モデル(式(1))を仮定する.観測数
は,推定誤差を無視できるほど十分に大きいとする.説明変数を
,応答変数を
として線形回帰したときの残差を
(6)
と表す.このとき,観測変数
が因果的順序において最初の変数になることができるのは,観測変数
が,残差
と独立であるときに限る.
この定理は,説明変数の因果的順序を決める上で,重要な役割を果たす.
2.2.3 2変数間の独立性を測る
それでは,次にLiNGAMの中で,説明変数と残差の独立性を相互情報量を使って測定することを考えてみよう.
変数 と残差
(7)
として,相互情報量は,
(8)
とエントロピー
を使って表現できる.
変数と残差
(9)
として,相互情報量は,
(10)
となる.
基本的には,これらの相互情報量の大小関係を調べるわけであるが,計算の前に,それぞれの変数を標準化して,標準化した量の変数の記号の上にバーをつけて表すことにする.たとえば,
(11)
などである.
相互情報量計算の前に,
(12)
と標準化したベクトルをつかって
(13)
と定義される量
を導入して,
なら
の方が
よりも因果的順序が早いと判断する.
2.2.4 DirectLiNGAMは因果的順序をどのように構成するか
変数間の独立性を測る方法を得たので,順序を構成することができる.複数の変数の因果的順序を求めるために,
(14)
という量を導入し,
が最大になる変数を因果的順序の最初になることのできる変数であると推定する.ここで,
は候補となる変数の添え字の集合である.
最初の変数が決まったら,その変数の添え字を から取り除く.そして,基本的には再び を計算したいのだが,今,親の変数を仮に とすると,残りのすべての変数に親変数 の寄与が含まれているはずであるから,ほかの変数については, で回帰した残差を親変数の影響が取り除かれた変数と思うことにし,再び を計算して残りの変数の親を決める.
今の例だと,ほかの変数というのは のことだから,
(15)
という計算で,親変数の影響を取り除き,
を
と更新して,
から
を再び計算して,観測変数
の親を決める.これを繰り返していくことで,最終的に,変数
が因果的順序で
番目であることが決定できる.
2.2.5 係数行列の推定
因果的順序 が推定できたら,今度は係数行列を構築する作業に入る.因果的順序で,自分より前にある変数すべてを説明変数として回帰し,そのときの偏回帰係数を係数行列の要素 とする.
すなわち,回帰モデルは
(16)
であり,ここで,は
(17)
で定義される,変数よりも因果的に前にある変数の添え字集合である.
基本的には,上の回帰式で直線回帰を行いたいのだが,説明変数の数が多い場合には,適切な正則化を行って回帰をしなければならない.清水らの方法では,ここでAdaptiveLassoを適用している.我々は,Elastic Netを用いてスパースな偏回帰係数を求めた.
3.大阪府民のKDBデータ
我々が今回解析に用いた国保データベース(以下,KDB)とは,国民健康保険団体連合会(以下,国保連合会)が保険者の委託を受けて行う各種業務を通じて管理する,国民健康保険(以下,国保)に加入している被保険者の健康記録や介護記録などの健康情報を保持したデータベースのことである.我々のプロジェクトは,生活習慣病等の予防と健康寿命延伸を目指した調査分析を目的としており,大阪府保険者協議会および大阪府国保連合会の協力によって,匿名化された大阪府民の国保加入者約200万人規模のKDBデータの提供を得ることができた.これらのデータを隅々まで把握し分析を行うためには,医療の専門知識を要することはもちろん,データハンドリングの能力も欠かすことはできない.それゆえ,筆者らは医療・数理統計・物理の研究者からなる研究グループを結成し,多くの分野の共同研究を行いながらプロジェクト推進を目指している.これからKDBデータを利用したデータ解析を行いたいと考える読者はもちろんのこと,筆者らにとってもこれほどまでのビッグデータを触れる機会というものはこれまでになく,合計で1テラバイトほどのテーブルデータをコンピュータでどのように扱ってきたかを医学物理を専攻してきた解析者の立場から記していきたい.
初めに大阪府民のKDBデータ(以下,大阪府KDBデータ)の概要を記述し,その後,大阪府KDBデータに触れて気付いたデータの問題点や注意点,実際に大規模な健診データに対してPythonを用いたデータ前処理や因果探索プログラムの実装について記載する.
3.1 大阪府KDBデータの構成
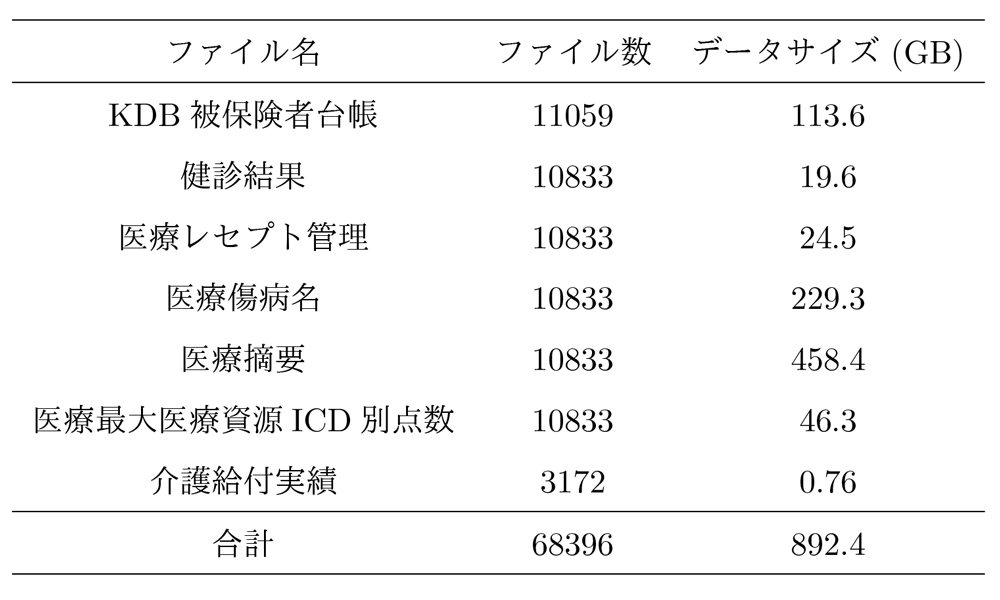
今回大阪府から提供を受けたKDBデータは,協力を得られた41市町村の2012年度から2018年度までの7年度分のデータが含まれており,大阪府KDBデータは以下の7種類のデータから構成されている.
- KDB被保険者台帳
- 健診結果
- 医療レセプト管理
- 医療傷病名
- 医療摘要
- 医療最大医療資源ICD別点数
- 介護給付実績
これらのファイルは大阪府の市区町村・処理年月ごとに1つのCSVファイルに分割されており,約68,000に分割されているCSVファイルをすべて合わせるとおおよそ1テラバイトの大規模なデータサイズとなる(表1).特にKDB被保険者台帳,医療傷病名,医療摘要は100ギガバイトを超えるデータサイズであり,これらを利用した解析を行う場合はデータ操作に注意しなければならない.筆者らは提供を受けてからデータの分析を始める前に,匿名化された個人番号を二重でハッシュ化した上で解析者に渡し,データファイルとヘッダ情報を別ファイルで保存しておく・使用するコンピュータを制限する・研究室からのデータ持ち出しの禁止などのルールを設けることで,セキュリティ対策にも細心の注意を払っている.
ちなみに,筆者はたまたま機会に恵まれて大阪府以外のKDBデータにも触れることがあったが,データベースの構造はKDBによって異なるようである.これは,KDBデータの場合,厚生労働省が全国レベルで一貫した管理が行われている「レセプト情報・特定健診等情報データベース(以下,NDB)」のデータとは異なり,市町村ごとにデータの管理や収集・構築が行われるため,作成した自治体によってはフォーマットなどが違ってくるのが理由だと考えられる.大阪府KDBデータは比較的整然とフォルダ分けされていた印象だったが,地域によっては(解析をするにあたっては)分かりづらい可能性もあるので解析前に必ず確認が必要である.
これらのファイル間は個人番号(ユニークID)で突合することが可能であり,解析に必要なファイルを適宜突合して解析に臨んでいる.本稿の解析では,これらのファイルのうち,主に被保険者の特定健診の結果が記載された健診結果ファイルを使用する.ただし,健診結果のファイルには被保険者の年齢と性別の情報が欠如していたため,これらの値は突合したKDB被保険者台帳から性別と生まれ年を抽出し,性別は被保険者台帳の値をそのまま利用,年齢については抽出した生まれ年と健診実施年月日との差分から計算し,これを利用した.
3.2 大阪府健診データのフォーマット
特定健診は,メタボリックシンドロームに着目して病気のリスクの有無を検査し,生活習慣病の予防を目的とした,40歳から74歳までの国民を対象に行われる健康診査である.75歳以上の場合は後期高齢者医療制度に加入することになるため,後期高齢者医療健康診査を受けることになり,特定健診の対象とは厳密には異なるが,本稿では特に区別することなくまとめて健診データと呼ぶ.なお,本稿の解析には単年の健診データのみしか利用していないが,縦断研究を行う場合は国民健康保険制度から後期高齢者医療制度への切り替えが存在することに注意しなければならない.
健診データには,身長や体重,腹囲などの身体計測,血圧を測り循環器系の状態を測定する血圧測定,中性脂肪やコレステロール値などの血中脂質検査,ALT(GPT)やAST(GOT)などの肝細胞障害の状態を測定する肝機能検査,空腹時血糖やHbA1cなど糖尿病の判定基準となる血糖検査などの項目が含まれる.また,脳卒中や心臓病などの既往歴や喫煙・飲酒,運動や食生活等の生活習慣に関する質問票データも含まれている.さらには医師の判断により実施された場合には,心電図検査や眼底検査,血清クレアチニンやeGFRといった血液検査の結果も記録されており,生活習慣病の発症リスクが高いと判断され,特定保健指導を受けた場合はそれらのデータも記録されている.そのほかに特定健診を実施した日付やメタボリックシンドローム因子の有無などの項目を含めると,全部で112の項目から構成されており,健診データを利用することで健診を受けた時点での被保険者の健康状態を取得することができる.
4.KDBデータを扱う際の注意点
KDBのような大規模データを研究に利用する最大の利点は,国保に加入した被保険者のデータを網羅した悉皆性の高いデータであり,対象者に偏りが少なく母集団の代表性に優れていることだと思われる.また,疫学研究でたびたび問題となるようなサンプルサイズも,余程稀なアウトカムを選択しない限りは問題とならないだろう.一方で,KDBデータを利用するにあたって非常に高い障壁となるのがデータハンドリングの困難さであり,研究の多くはデータの理解とクレンジングが大半の時間を占めることになると予想される.
ここでは,研究チームでの議論の中で得られた考察や,実際にデータを触ってみてようやく気付いた問題点などを筆者の経験に基づいて記載する.ここで記載した内容が初めてKDBデータを触る人たちの一助となれば幸いである.
4.1 被保険者数の数合わせ
大阪府KDBデータに何人の被保険者が存在するかを確認するため,筆者らは最初に被保険者数の数合わせを行った.まず,2012年度から2017年度までのすべての被保険者台帳のデータ(以下,台帳データ)からハッシュ化された個人番号を引き出し,重複を除いた人数をカウントした.さらに,ある特定の日に被保険者がどれくらい存在しているかをカウントした.筆者らはこれらの作業を数名の解析者が独自のプログラムで単独で行い,それぞれ算出した人数を突き合わせることでこれらが一致するかを確かめた.データをいただいた当初は膨大なデータ量と慣れないデータ形式から人数が大きく異なっていた.特に,台帳データの項目の意味を読み解く作業にほぼ1年を費やし,その間に台帳データそのものに不足分があることも判明した.初めは比較的人口の少ない市町村のみでの数合わせを行いながらKDBデータの癖とコーディング手法を学び,台帳の不備を補った後,さらに1カ月ほどの時間を費やして,最終的に解析者全員の個人番号の数が大阪府全体で9,421,332名で一致した.大阪府の人口が2021年度現在で大体900万人であるため7年間の台帳であることを考慮しても随分多いように思えるが,これはKDBデータの特性であり,国保から後期高齢制度に移った場合は追跡ができず,重複してカウントしてしまう,国保の場合は転居などで市町村を移動した場合は個人番号が変わってしまうといった理由が存在するためである.追跡調査を行う場合は,このような点にも注意しながら解析しなければならない.なお,台帳に含まれる人数をある一時点でカウントした場合は,どの年度でもおおよそ200万人ほどであった.
続けて,台帳と同様に2012年度から2018年度までの全データベースを対象にした個人番号のカウントも行った.両者の個人番号の数が一致することが確認できれば,被保険者台帳に含まれていないにもかかわらず,健診やレセプトデータなど,そのほかのファイルを持つ被験者が存在する,といったおかしなデータがないことが確認できるからである.この結果,両者の数値は一致し,台帳に含まれていないが健診データが存在するといった人は大阪府KDBにはいないことが確認できた(台帳には存在するが,そのほかのデータを持っていない被保険者はたくさん存在する).
4.2 性別と生年月日の不一致
被保険者の人数が解析者間で一致したため,数合わせについてはこれで一件落着と思われた.だが,続けて個人番号と性別・生年月日の関係を調べた際に新たな問題が生じた.台帳ファイルには被保険者の性別と生まれ年の情報が記載されているが,同一の個人番号にもかかわらず,性別や生まれ年が異なる被保険者が含まれていたのである.全体の人数と比較すると大した数にはならないが,性別が一致しない被保険者は227名,生まれ年が異なる被保険者は57名存在した.台帳データの更新時に入力に誤りがあったのか,同一の個人番号がたまたま振られてしまったのか,理由を特定することは難しいが,いずれにせよ性別や生まれ年(年齢)はどのような解析を行うにしても非常に重要な因子となり得るのでこれらのデータを持つ被保険者は全解析から除き,残った9,421,051名を解析の候補とした.
4.3 保険加入期間の定義
今回提供を受けたKDBデータは国保加入者を対象としている.そのため,台帳データには被保険者ごとの「国保(後期)取得年月日」と「国保(後期)喪失年月日」が記録されており,被保険者が国保に加入していた期間を定義する必要がある場合にはこれらの項目を参照した.何らかの理由で国保の喪失があった場合でも再度取得していれば,特に問題がない限りは台帳には必ず記載されている.ただし上述したように,KDBデータでは市町村が変わると追跡できないので注意する.同様に介護資格の取得/喪失年月日も被保険者台帳には含まれており,介護認定を受けた期間などもここから取得可能である.余談ではあるが,有効期間の開始日と終了日という項目が台帳には記録されているが,これらが示す日付の定義が分からなかったため,現在はどの解析にも利用していない.
4.4 傷病名の定義
続いて目的変数や介入の有無などに傷病名を利用したい場合について言及する.KDBには診療報酬明細書である医療レセプトデータが含まれており,患者の傷病名と行われた医療行為が記録されている.そのため,疾患をアウトカムとするような研究デザインを計画する際には,医療傷病名データに含まれる傷病名やICD-10を利用したいと考えたが,研究チームでの議論の中で,医師からはこれらの利用には慎重になるべきとの指摘があった.
当時,筆者は知らなかったが,レセプトデータには疑い病名と呼ばれ,レセプトで検査や投薬を行うためには病名が確定していなくても,病名が疑われる状態で病名を記入しておくことで検査や投薬を実施するという慣習があるようだ.たとえば,鎮痛薬を使用する患者の胃炎防止のために胃薬を処方したい場合,実際に胃炎に罹患していなくてもレセプトに胃炎の病名を記載する,ということがあるそうだ.このような問題は厚生労働省が公表しているNDBを利用した解析でも議論されており,レセプトデータに記載された傷病名やICD-10の妥当性の調査を目的としたバリデーション研究なども行われている.もちろん,ICD-10を実際に利用するかどうかはどの傷病を対象にしたいかにもよるだろうし,研究チームの方針にもよるだろうが,どうしてもこの慣習が研究利用の足枷となってしまう.筆者らの研究チームでは,レセプトデータの傷病名は極力利用せず,その代わりとして医療摘要データに含まれる薬効分類(ATC分類コード)を利用し,実際に処方された薬から傷病名を定義している.
4.5 KDBデータの項目をそのまま利用すべきか
続いて,KDBデータの値をそのまま鵜呑みにすべきでない場合があることを筆者の犯したミスと経験に基づいて言及する.大阪府のメタボリックシンドロームの罹患率を調査する目的で,筆者が被保険者のBMIから肥満と定義される群の人数のカウントを行ったときのことである.BMIは健診データに記録されており,年齢と性別で層別化してそれぞれの群で肥満の割合を算出していたが,ここで筆者のミスに気が付いた.日本肥満学会の定めた基準では,「BMIが25以上」の群を肥満としているところを,「BMIが25より大きい」群を肥満としてしまっていた.しかし,よく知られているように,BMIは身長と体重から算出される連続量であることから,解析にはほとんど影響しないだろうと考えていた.だがその予想は大きく外れ,肥満群に含まれる人数が数千人単位で変化してしまったのだ.当時筆者は驚いてしまったが,聡明な読者の方々であれば簡単に気付く自然なトリックであり,その大きな原因はBMIが小数点以下第一位までしか入力されていなかったためである.つまり,実際にはBMIがピッタリ25でした,という人はほとんどいないだろうが,四捨五入が行われたせいでBMIがちょうど25になってしまった人が思いの外多かったのである.きっかけは筆者の些細なコーディングミスであったが,本稿の因果探索を始め,BMIなど別の値から目的の変数を算出できる場合は,両者を比較して適切な方を利用することを決まりとするようになった.
これは糖尿病診断基準など,上の傷病名の定義にも通ずるものであり,傷病名と検査項目の両方から定義できる疾患や,質問票・既往歴・薬効分類などから網羅的に特定できる疾病についても,どの項目を利用するか,あるいは複数の項目で1つでも当てはまれば疾患と見なすなど,医師や疫学者を交えて慎重な議論が必要となる.
4.6 健診データのクレンジング
ここでは,健診データの前処理方法について記載する.KDBデータに限らず,多くの医学データや健診データは日々の診断や治療,介護の状態を記録したものであり,データ解析に扱いやすいよう整えられているものではない.そのため,これらのデータを解析に用いるために,各種解析ソフト(Python,R,SASなど)で扱いやすい形に成形してやる必要がある.この作業をデータのクレンジング(クリーニング)と呼ぶ.
まず初めに,KDBに含まれる全健診データのうち,条件抽出で2016年度の健診データのみを呼び出す.今回,2016年度を解析対象とした理由は,各年度の健診データの中で最も被保険者数が多い年度であったためである.この時点で重複した個人番号を除いた結果,679,351名の被保険者が解析対象の候補となった.また,本解析では多重共線性の影響を考慮した上で,健診データから以下の11変数を解析に使用した.
- 身体計測(身長:height,BMI)
- 血圧関係(収縮期血圧:sBP)
- 脂質関係(LDL コレステロール:LDL,HDL コレステロール:HDL,中性脂肪:TG)
- 肝機能関係(GOT,γGT,GPT)
- 血糖値関係(空腹時血糖:fBG,HbA1c)
ただし,上でも述べたように,BMIだけは健診データの身長と体重から算出した.次に,これらのデータをLiNGAMモデルに投入できるよう健診データに欠損を含む被保険者を解析対象から削除する.ここで気を付けなければならないのは,特定健診の項目や所属する市町村によって欠損の定義が異なる,ということである.表2に使用した変数の基本統計量を示す.
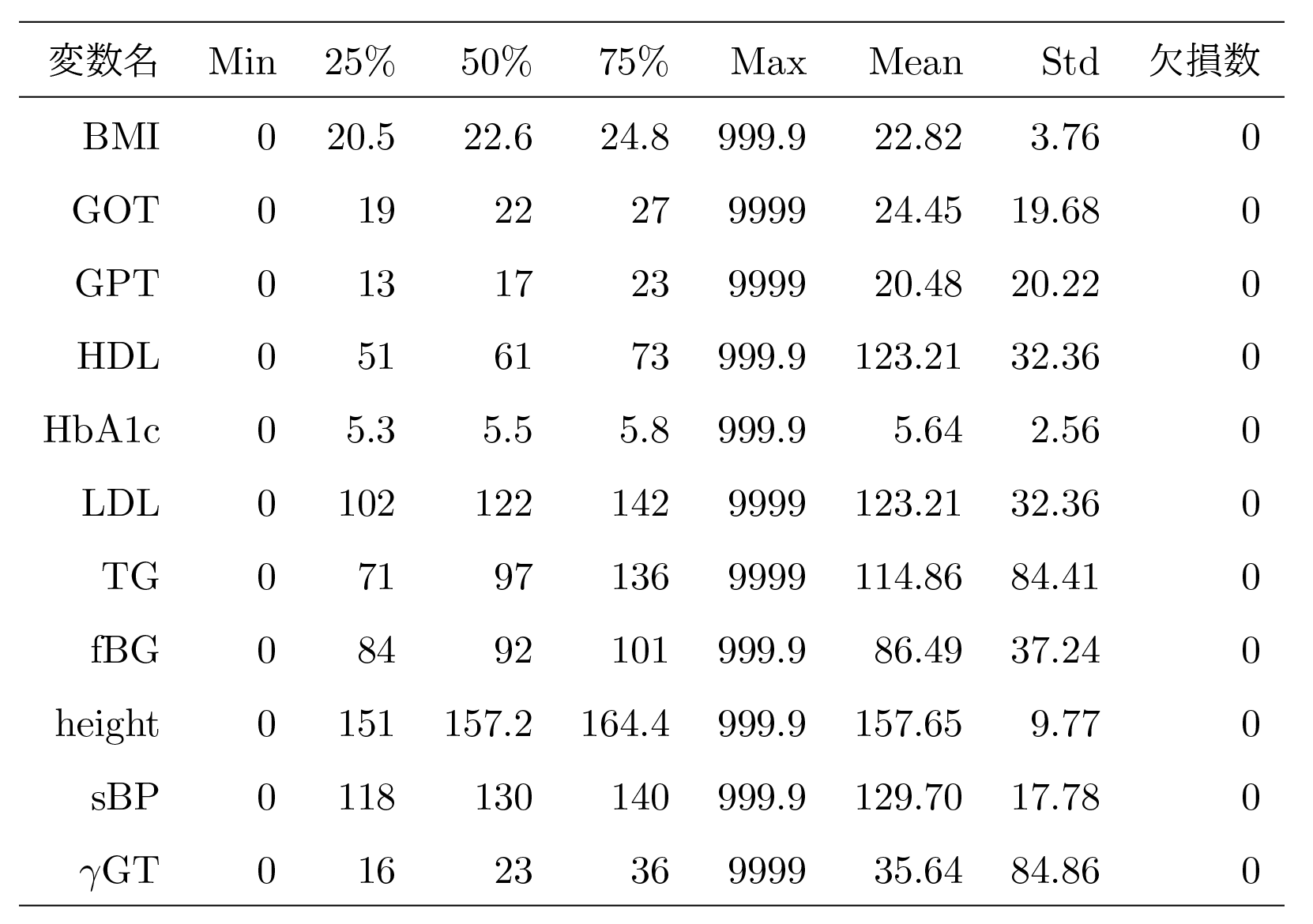
表2は679,351名から得られた統計量である.以下の章「Daskによる分散処理」にてDaskと呼ばれるPythonのライブラリを使用した欠損値の削除方法の例を記載するが,dropnaで削除可能な欠損値はブランク(空欄)のみである.表2から見てとれるように,使用したすべての変数はブランクを含んでおらず,上記の方法で削除可能なデータは存在しない(ただし,血清クレアチニン検査値やeGFRなど,検査項目によってはブランクを含むデータが存在することに注意しなければならない).しかしながら,各変数の最大値と最小値に着目すると,最小値はすべての値で0であり,最大値は変数によって異なるが,999.9や9999といった通常の検査範囲では取り得るはずのない値が入力されていることが見てとれる.これらの値は,健診データの入力者が便宜上何らかの理由でブランクの代わりに代入したものだと想像できる.
それゆえ,我々は欠損値を削除する代わりに,健診時に取り得るはずがなく,かつ人為的に入力されたと考えられる値を異常値と定義し,これらの値の削除を行った.加えて,異常値とは別に,入力者のタイピングミスによって想定し得る検査値から大きく外れた値を持つデータも存在した(たとえば,身長が16.8cmと入力されているデータが存在した.これは168cmのタイプミスだと容易に想像がつくが,こういったデータを意図的に修正すべきではない).安定した解析結果を推定するために,これらの値を外れ値と見なして削除する必要があった.
5.Pythonを用いたビッグデータ処理
近年ではビッグデータが注目を集めており,KDBデータに限らず,膨大かつ多種多様なデータをどのように管理し,データの処理や分析を行うかが問題となってくる.大規模データのハンドリングに関してはエンジニアに委託する手もあるが,アカデミアの学術研究として研究者ら自身でデータの管理や成形ができるに越したことはない.
一般的な表形式の大規模データの分析手順をまとめると,Hadoopなどの分散処理技術によって分割されたビッグデータの格納・処理を行い,SQLなど収集したデータを整理・操作するためのデータベースの形式に落とし込み,必要なデータを条件抽出することによってようやく各種分析ソフトで扱えるようなデータになる.現在は多くの参考書が出回っており,ビッグデータ解析のためのセミナなども開催されているためこれら技術の習得はそこまで難しくはないように思える.しかし,各ソフトウェアの理解や習得には膨大な時間を要することと,筆者らのチームが汎用性の高いプログラミング言語であるPythonを主に使用してきたことから,これらの処理をすべてPythonのみで実装することにした.
Pythonは人工知能やデータサイエンスが注目を集める中で最も人気を集めているプログラミング言語の1つであり,汎用性の高さと無料のオープンソースである点から研究開発から商業利用まで幅広く利用されている.
ここで,PythonでCSVファイルのような表形式のデータの処理や解析を行うための代表的なライブラリの1つにPandasが挙げられる.Pandasは表形式データのデータ解析を実施するための機能を提供しており,SQLやRのようなデータフレーム処理をPython上で可能とするライブラリである.Pandasはデータ解析を行うためのメソッド(実装済みの関数)が大変充実しており,Pythonでデータ解析を行ったことがある方やPythonの入門書を読んだことがある方は一度は目にしたことがあるのではないだろうか.
このように,Pandasは表形式データにおいて非常に便利なライブラリであるが,一方でビッグデータ解析には向かない面も存在する.Pandasはオンメモリでの解析を前提としているため,Excelなどでも開くことができるCSVファイルであればなんの問題もないが,大阪府健診データのようなビッグデータをPandasで扱おうとすると,コンピュータの処理が非常に重くなるか,動作中にメモリエラーとなり,そもそもデータを扱うことができなくなる可能性がある.このような問題はPandasに限った話ではなく,RやSAS,SPSSといった解析ソフトでも同様のエラーが生じ得る.解決策として,大容量のメモリを搭載したPCやワークステーションの購入を検討すべきであろうが,予算の都合もあるだろうし,今後膨大に増え続ける可能性が高い健診ビッグデータに対してマシンパワーにコストを費やし続けるのは最善策とは言い難い.
そこで我々は,Pandasでは扱いきれないような大規模なCSVファイルに対して,並列処理や分散処理を容易に行うことのできるDaskというPythonライブラリを紹介する.Pythonを使ってKDBデータ処理を検討されている読者の参考になれば幸いである.
5.1 Daskによる分散処理
Daskは並列処理や分散処理を用いて,一般的なコンピュータではメモリ不足となるようなデータサイズの大きなデータセットに対しても処理や解析を実施するためのライブラリである.ビッグデータを扱うためのPythonライブラリはほかにもいくつか候補が挙げられるが,DaskはPandasのコーディング表記に非常に類似しており,またDaskとPandasのデータは相互に変換可能であることからデータ解析に不慣れなPythonユーザでも取っ付きやすいだろう.Python上でDaskを使用するためには,pipコマンドでインストールするか,筆者のようにAnacondaでPython環境を構築した場合は,最新のAnacondaをインストールすればすでにインストールされているはずである.
たとえば,Pandasを用いてCSVファイルを読み込む場合は下記のように記載するだろう.
df = pd.read_csv( 'data.csv' )
一方でdaskを用いてデータを読み込む場合は下記のように書き換えればよい.
ddf = dd.read_csv( ' data.csv ' ,blocksize=10e6 )
上記のように,ほとんどPandasと同じような表記でCSVデータを読み込むことができることが分かる.ここで,blocksizeは1つのパーティションのデータサイズを指定しており,上の場合は1パーティションあたりの最大データサイズが100メガバイトとなるように自動で分割してくれる.Daskでの処理はこのパーティション単位で行われ,適切なblocksizeを指定することで並列処理を自動で行ってくれる.分割したいパーティション数を直接指定することも可能で,この場合はrepartitionのメソッドでnpartitionsに分割数を指定すればよい.
上の場合は,パーティションが8つになるように自動で分割してくれる.特別,並列処理を行うためのコードを記述せずとも,CPUが許す限りは並列で実行してくれる.
次にDask上でデータ処理を行いたい場合,たとえば欠損のあるデータの削除を行いたいとする.Pandasではdf.dropnaと記載するが,Daskでも同様の記法で実行できる.
ddf = ddf.compute( )
このように,Pandasで実行可能なメソッドの大半はDaskでもほぼ実行可能だと思って差し支えない.また,Pandasの利用に慣れている場合は,メソッドチェインを用いて複数の処理を1行で書くことも可能である.一方でDaskでは,dropnaと記述したコードを実行した時点で即座に欠損値削除が行われるわけではない.Daskを用いて記載した処理は内部で記録され,最後にcomputeと入力することで初めて処理が実行される.なお,最終的にcomputeで渡される変数はPandasのデータフレームとなるため,compute以降はそのままPandasを用いたデータ解析や可視化手法等を行うことができる.
ちなみに,Dask内でどのような並列処理が行われているか確認したい場合は,computeの代わりにvisualizeと入力することで図4のように内部の処理過程を可視化することもできる.
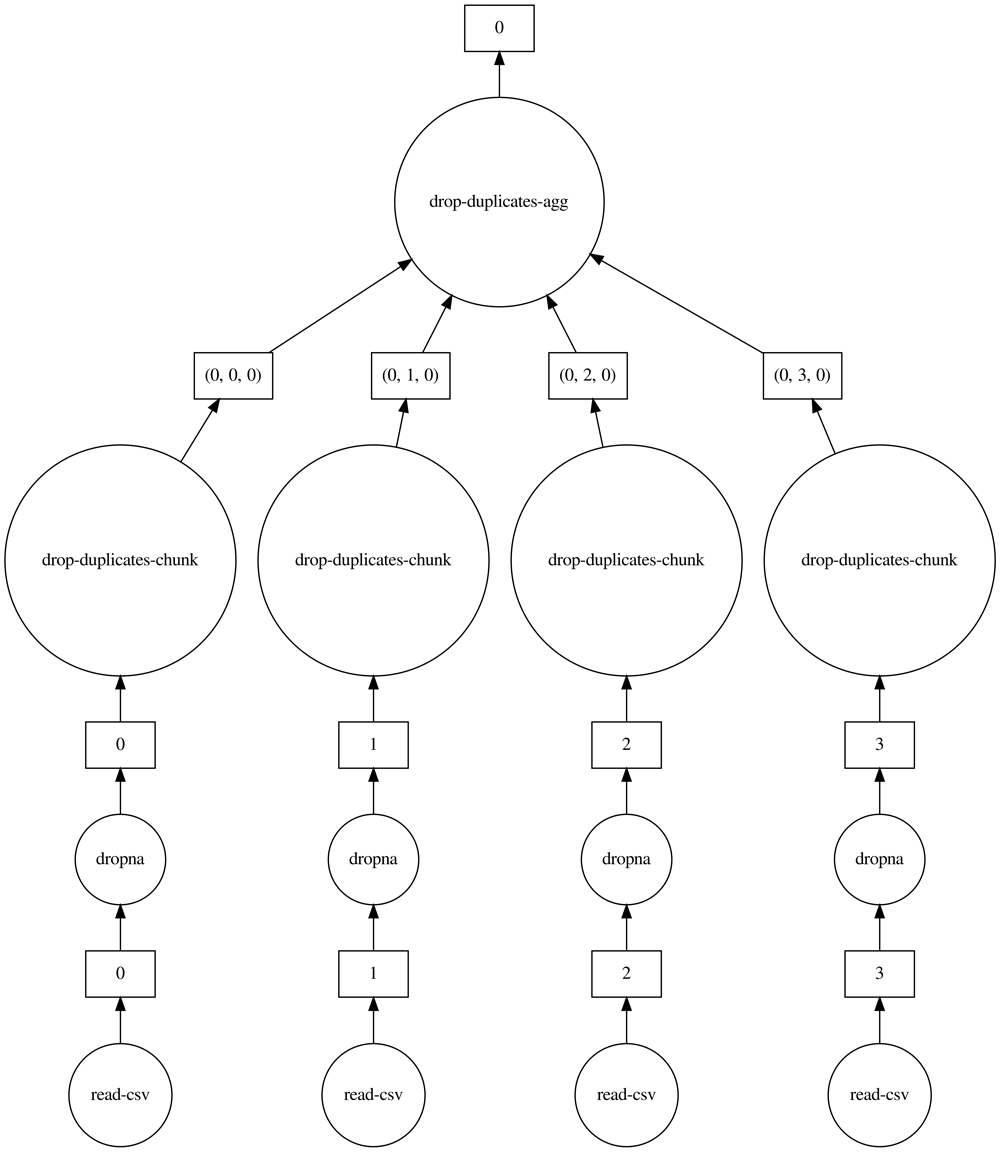
ほかにもDaskを利用するメリットとして,複数ファイルを読み込む際にもPandasと比べて簡潔に記載することができる.下記のように,Pandasのread_csvは1つのファイルパスしか指定できないため,複数のファイルを読み込むためにはfor文などで1つずつ呼び出す必要がある.
'data2.csv ' ,
'data3.csv ' ]
dflist = [ ]
for filename in filelist :
df = pd.read_csv( filename )
dflist.append( df )
df_all = pd.concat( dflist )
一方でDaskではファイルパスのリストやglob string(ワイルドカード)を含んだファイルパスを使用することが可能である.
'data2.csv ',
'data3.csv ']
ddf_all = dd.read_csv ( filelist ,blocksize=100e6 ) . compute ( )
Pandasで作成したdf_allと,Daskで作成したddf_allは,reset_indexなどのメソッドでインデックス名を振り直せば同じ出力結果が得られる.年月や市区町村で細かく分割された健診データをfor文を使わずに読み込める利便さを考えると,大きな利点である.詳しい操作方法についてはDaskの公式リファレンスを参照にしていただきたい(https://docs.dask.org/en/latest/).
ただし,DaskはNumpyやPandas,Scikit-learnのようなAPIを完全にサポートしているわけではないため,メモリ不足となりやすいデータの突合やデータクリーニングにはDaskを使用し,中小規模のPCでも扱えるほどのデータサイズまで落とした後はNumpyやScikit-learnを利用するのがよいだろう.また,データの突合の際など,パーティションを分割しすぎると計算時間がかかりすぎてしまい,かえって不便になってしまう場合がある.その際にはパーティションの数を見直したり,ある程度の処理ごとにcomputeして中間ファイルを作成するなどの対策を取るとよい.
5.2 異常値と外れ値の処理
本解析データに欠損値(ブランク)が存在しなかったため,データの前処理として異常値の削除と外れ値の削除を行う.ここで,異常値とは,前述の通り実際の検査範囲では取り得るはずがなく,かつ人為的に入力されたと考えられるデータのことである.これは表2の0や999.9,9999などが該当する.まずはこれらの値を定義しなければならないが,どの変数にどのような異常値が含まれているか把握できるまでは,表2のように変数ごとの最小値と最大値を記録し,異常値が含めれていたら抜く,という作業を繰り返しながら目視で確認する必要がある.とはいえ,普段から健診データを扱う機会の多い医師や保健師であれば数値を見ただけで異常値の定義は明らかであろうが,筆者のように自身の健康診断の結果しか見ることのない解析者にとっては,ただでさえ扱いの難しい大規模データであるのに加えてなかなか骨の折れる作業であった.最終的には医師などを交えて議論し,現在はデータの更新が行われるまでは記録しておいた異常値のリストを解析のたびに引用することにしている.
今回解析に使用した11の変数において異常値の割合を調べたところ,各変数の異常値の割合はfBGを除けば0.004〜0.51%と非常に小さく,HDLとGOTの異常値の割合が0.004%と最も低かった.一方でfBGだけは13.3%とほかの変数と比べて異常値の割合が随分と高かったが,これは,fBGは空腹時の血糖値を測定するため,食事をしてきた健診受診者の値は入力されないからだと考えられる.
異常値をすべて削除した後は,検査範囲から大きく外れた値(外れ値)を削除する必要があるが,外れ値の削除方法はすでにさまざまな方法が提案されている.筆者らは非正規分布の変数に対して利用されることの多い外れ値の削除方法として,各変数分布の上下数%のデータを外れ値として除くことにした.ここでは上下0.05%を外れ値の閾値と見なした.これらの処理をした後の基本統計量を表3に,各変数のペアプロットを図5に示す.
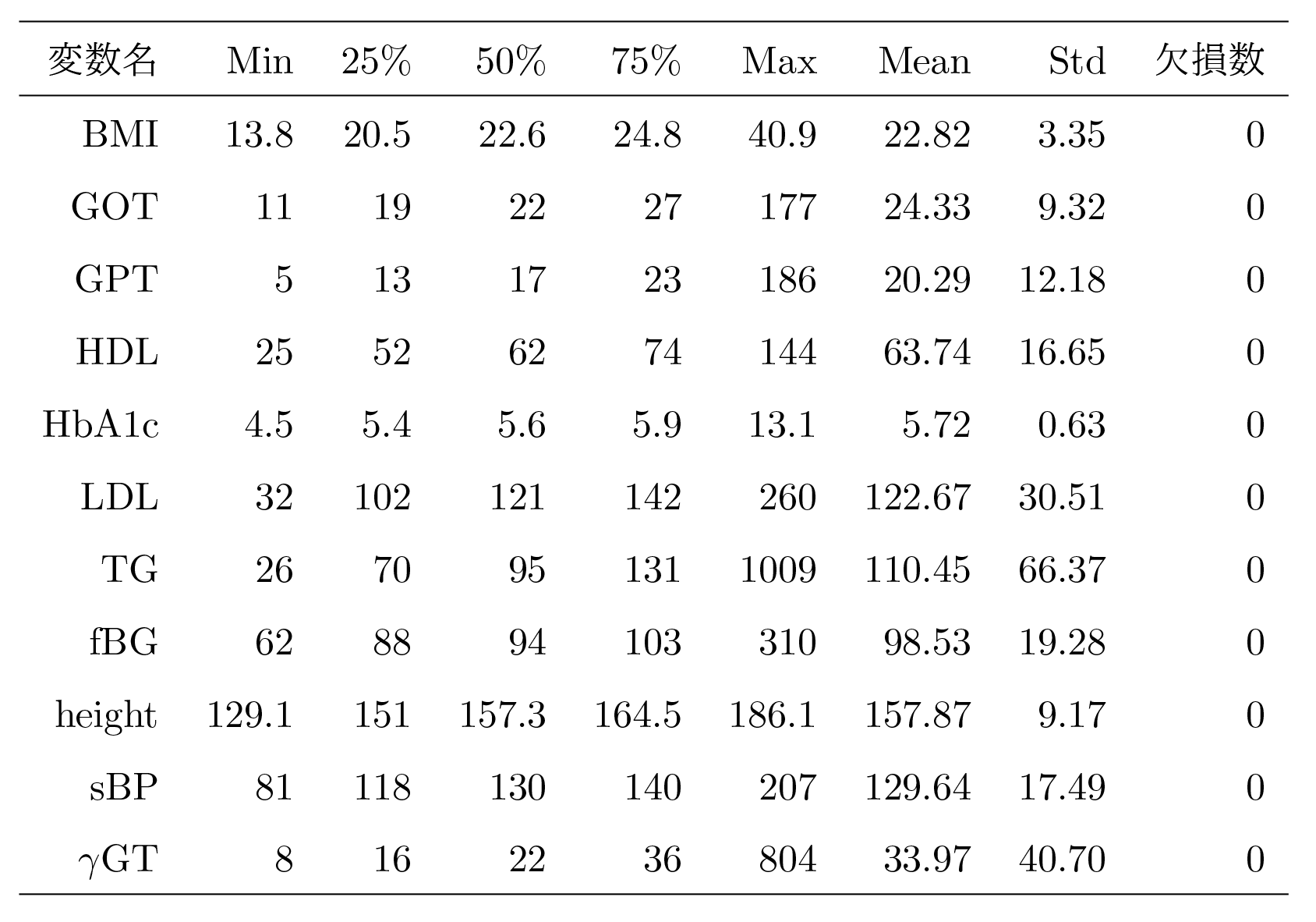
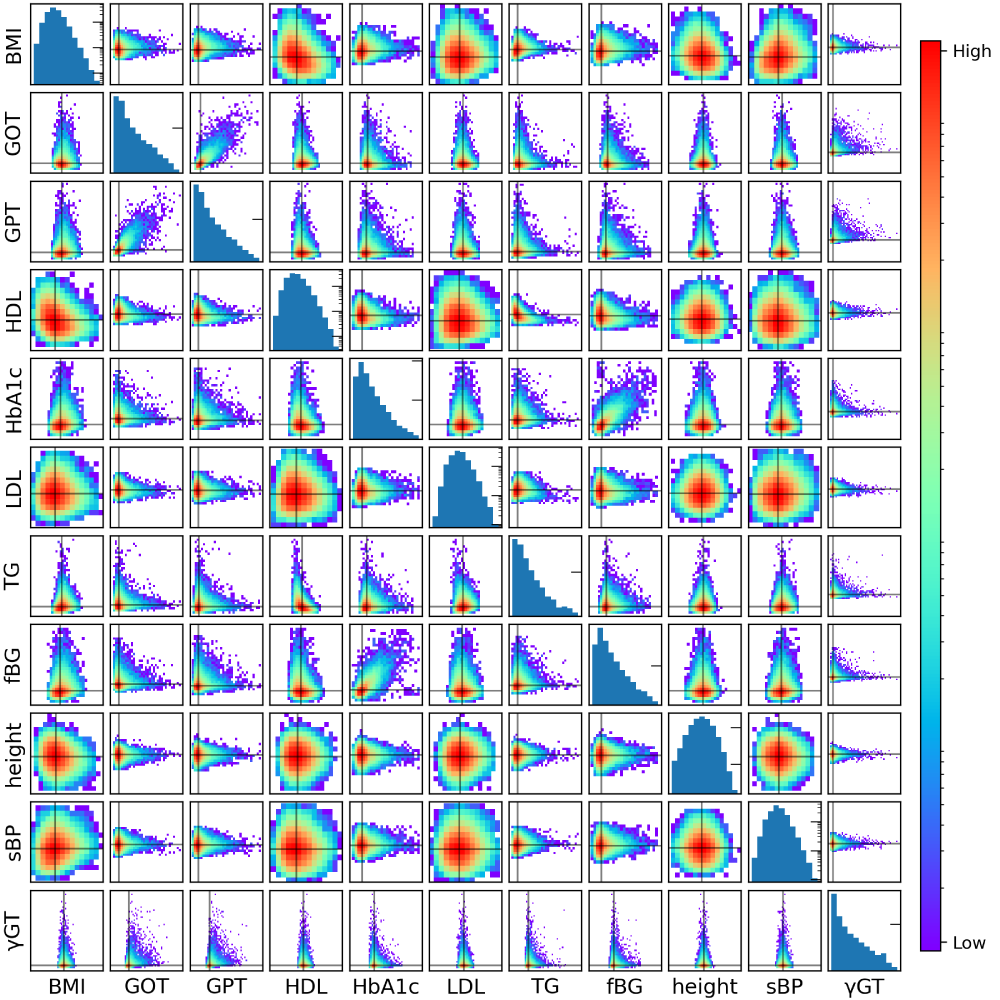
表3と図5は最も人数の多かった70代女性(N=131,036)の基本統計量と変数間のペアプロットである.表3を確認すると,異常値と外れ値を削除することによってもっともらしい統計量を得ることができた.同様に図5のペアプロットからも明らかな異常値や外れ値が含まれていないことが見てとれる.以降では,データ数の最も多い70代女性の解析データに焦点をあてて因果ダイアグラムを推定結果を示す.
6.大阪健診データを用いた因果ダイアグラムの推定
データクリーニングを終えた検診データを用いて,いよいよ因果ダイアグラムを推定する.前述の通り,DirectLiNGAMによる因果ダイアグラムの推定は,1.因果順序の推定,2.係数行列の推定の順に行う.
なお,性別と年代によって健康に対する指標は大きく異なるため,因果ダイアグラムを推定する際には男女で分割し,さらに年齢も10歳刻みで分割してから推定を行った(表4).
6.1 健診データを用いた因果的順序の推定
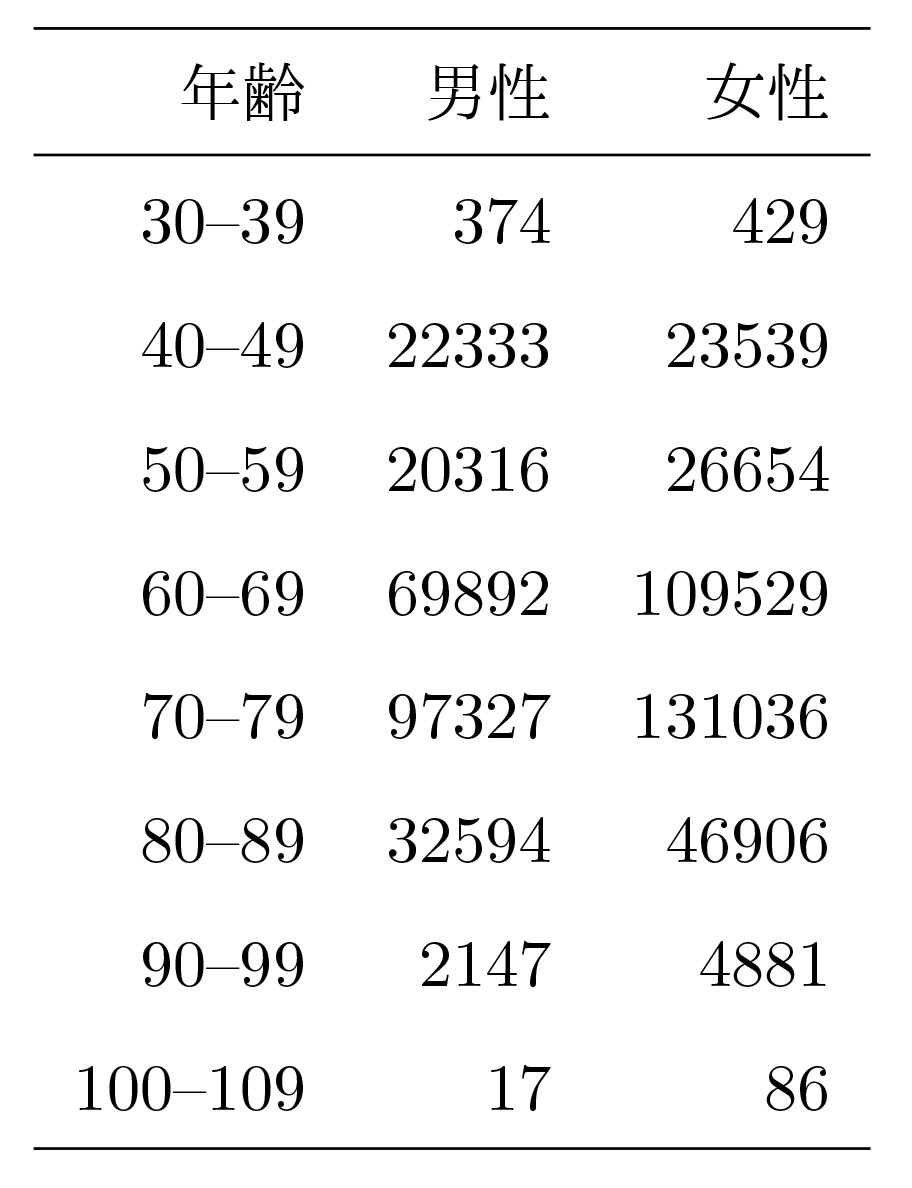
初めに,2変数間の独立性の測定を繰り返すことで,因果的順序を一意に推定する.例として,HDLとBMIの関係を調べてみる.図6の左上の図はHDLを横軸に取り,BMIを縦軸に取った密度プロットである.この図を見ると,HDLとBMIは負の相関を持っていることが見てとれる.これに対してHDLを説明変数,BMIを目的変数として線形回帰を行い,HDLに対して残差を2次元密度プロットで表現したものが右上の図となる.この図上で,線を引いた位置で断面図を取ったものが左下の図であり,それをそれぞれ規格化して条件付き確率の分布としたものが右下となる.これを見ると,3カ所で確認した例ではあるが,HDLとBMIは,かなり独立性が高いことが見てとれる.つまり,HDLはBMIの原因となっていると推論することができる.
上記の手順をすべての変数の組合せで行うことで,初めに因果的順序が最も早い変数を決定する.ここで,我々は解析を進める中で,因果的順序の推定に使用するサンプル数が比較的少ない場合,順序の推定にばらつきが生じることを確認した.そこで,復元抽出ブートストラップ法を用いて1,000個のブートサンプルを作成し,1,000回の因果的順序の推定を試みた.図7に最も早い因果的順序を決定するために計算した式のの推定結果を示す.
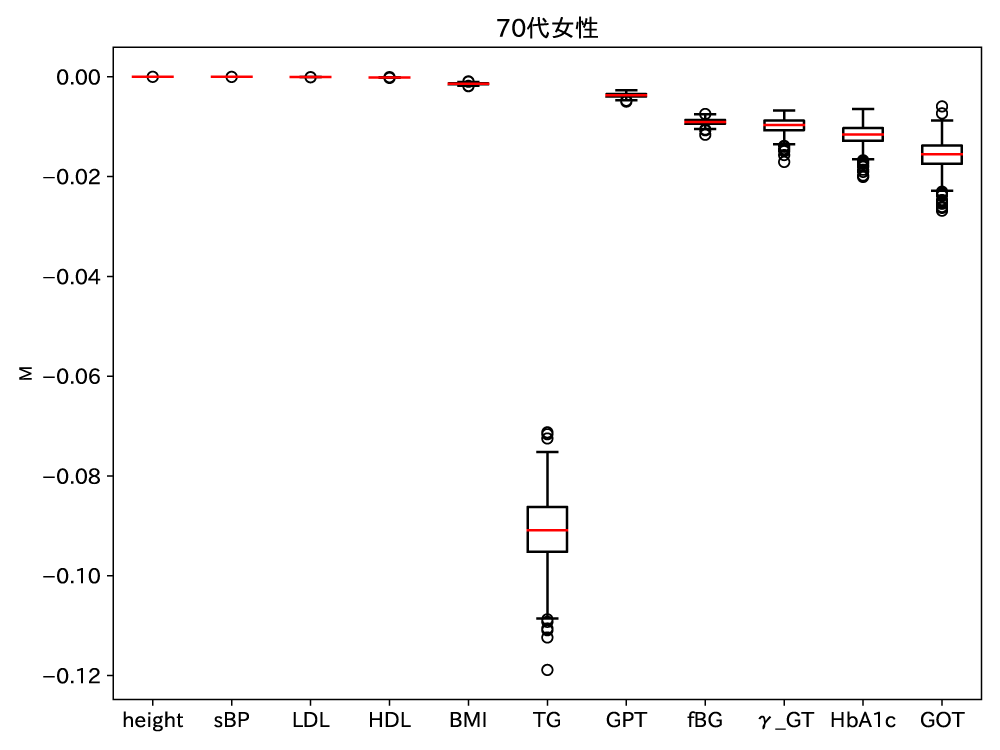
図7は1,000回のの推定結果を箱ひげ図でプロットしている.この図を見ると,height,sBP,LDL,HDLはほとんど違いがなく,どれも独立性が高いことが見てとれる,次点でBMIのの推定値が高く,それ以降はの値が小さくなっている様子が見てとれる.このような独立性の評価を繰り返し,最終的にブートストラップ法を用いて得られた1,000個の因果的順序を表5に示す.
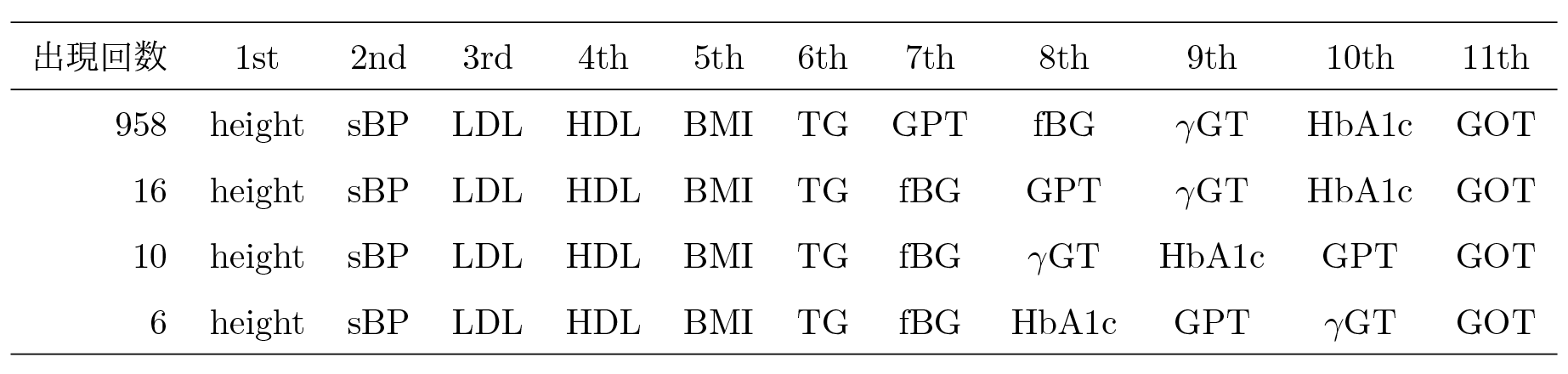
得られた1,000個の因果的順序はいずれも非常に似たような順序を示していることが見てとれる.因果的順序の早いものから順に,height,sBP,LDL,HDL,BMI,TGはどのパターンでも一致しており,身長や収縮期血圧,コレステロール値といった項目はどの変数からも影響を受けにくいことを表している.一方でHbA1cやfBGの糖尿病の指標となる変数や,GPTやGOT,γGTといった肝機能障害の指標となる変数は因果的順序が遅く,これらの変数はほかの変数から影響を受けやすいことが見てとれる.
なお,サンプル数の多い70代女性の場合,表5が示すように非常に頑健な因果的順序の推定を行うことができたが,サンプル数が少ないほかの年齢性別群で因果的順序の推定を行った場合は望ましい推定結果を得ることができない群もあった.望ましい因果的順序の推定を行うために必要となるサンプル数を調べるため,10,000から100,000人の間でランダムに解析対象者を抽出し,1,000回の因果的順序の推定の中で,最も多い順序のパターンが何回出現したかを記録した(図8).
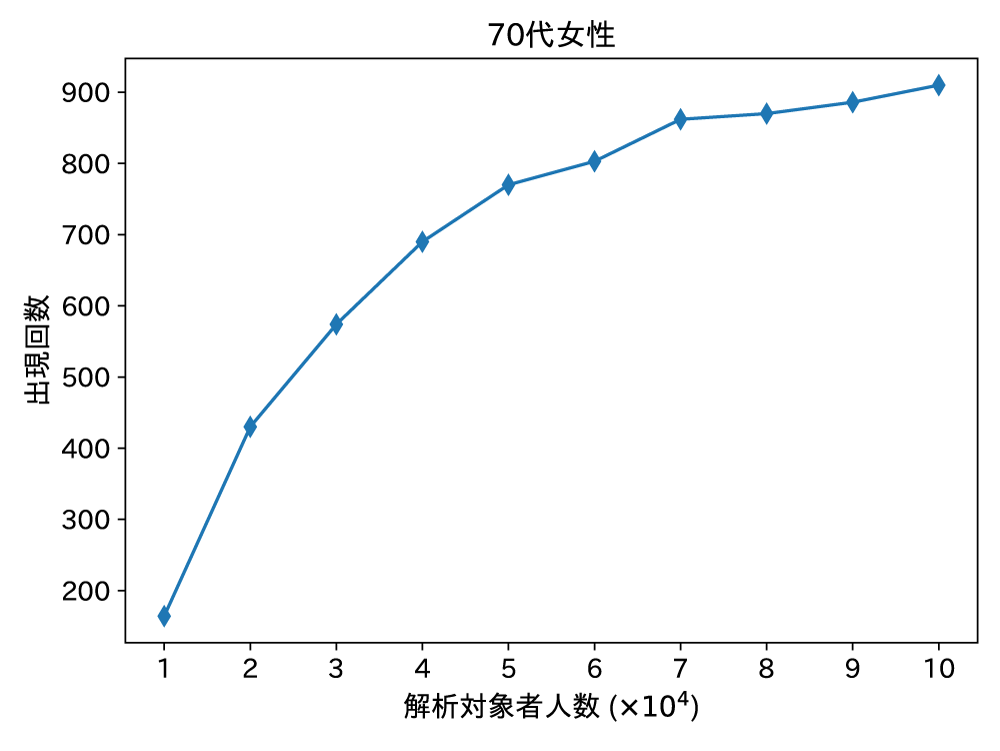
70代女性のように,もともと100,000を超える対象者を用いて因果的順序を推定した場合は,表5で示したように最も多いパターンで9割を超えていた.一方でサンプル数を小さくしていくと最頻のパターンはどんどん少なくなっていき,10,000名での解析では,最頻パターンでも2割に満たない結果となった.本解析に使用した変数項目の場合,おおよそ30,000を超えるサンプル数を用意することで,最頻パターンは5割を超え,満足な推定結果を得ることができた.
6.2 係数行列の推定
上で推定された因果的順序に基づいて係数行列を推定する.推定された因果的順序から回帰モデルを作成する流れは図9のとおりである.因果的順序のうち,自分(目的変数)より前にある変数すべてを説明変数とした回帰モデルを作成し,このモデルから得られた偏回帰係数を係数行列の要素とする.これを最も因果的順序が早い変数を除いたすべての変数が目的変数となるまで繰り返し,係数行列を作成する.
本稿の解析では,回帰モデルにElastic Netによって正則化を行った回帰モデルを使用した.さらに,推定された偏回帰係数のうち,因果関係の結びつきが弱い因果効果を削除(枝刈り)するため,因果的順序の推定時と同様に,係数行列を1,000回推定し,各係数の95%信頼区間を算出した.この信頼区間の中に0が入っていれば,その因果関係の結びつきは弱いと判断し,0を推定値とした.それ以外の場合は1,000回の推定値のうち,中央値をその因果関係の推定値とした.70代女性の健診データから推定された係数行列(表6)と,枝刈り後の係数行列から作成した因果ダイアグラム(図10)を示す.
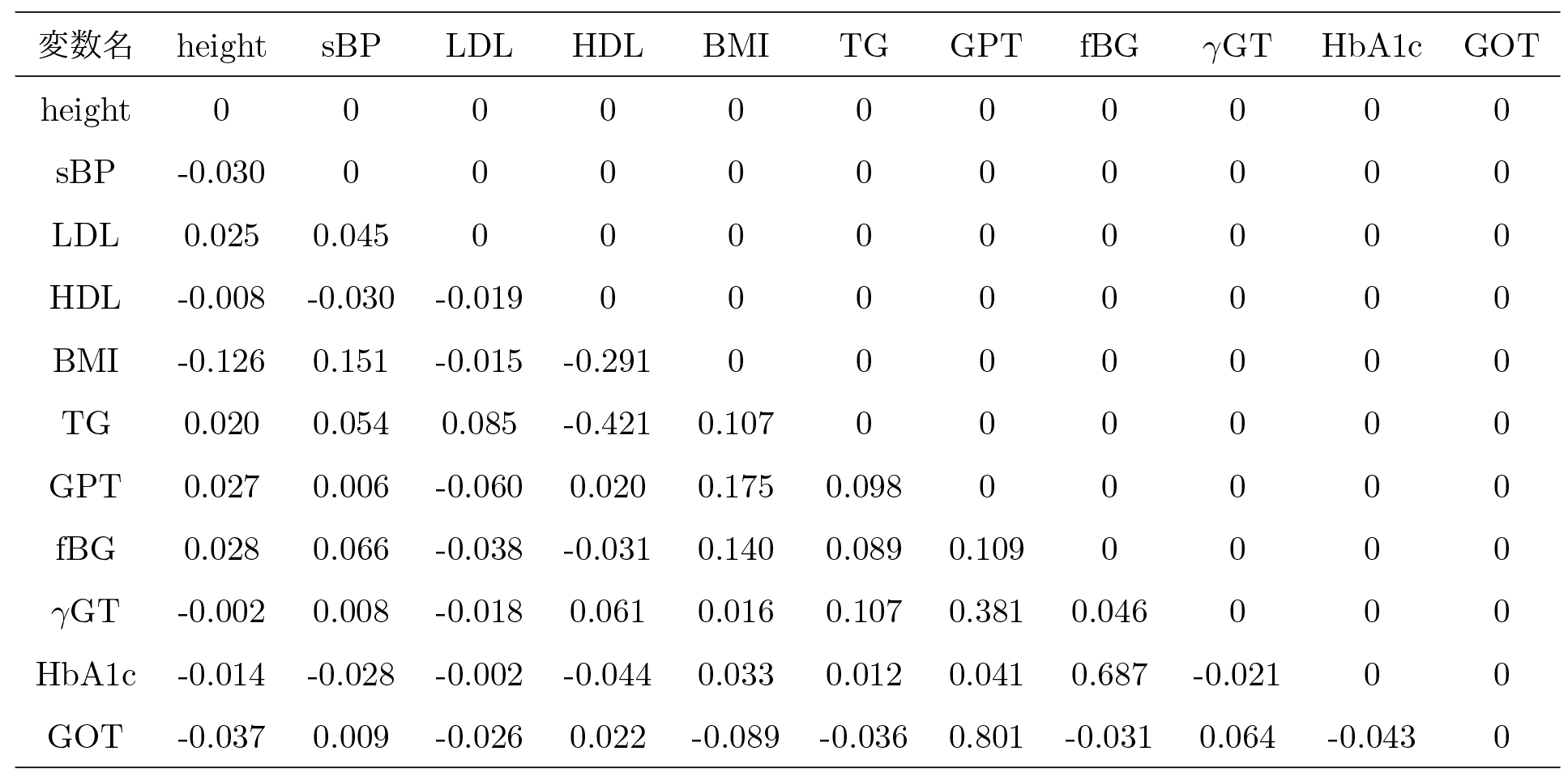
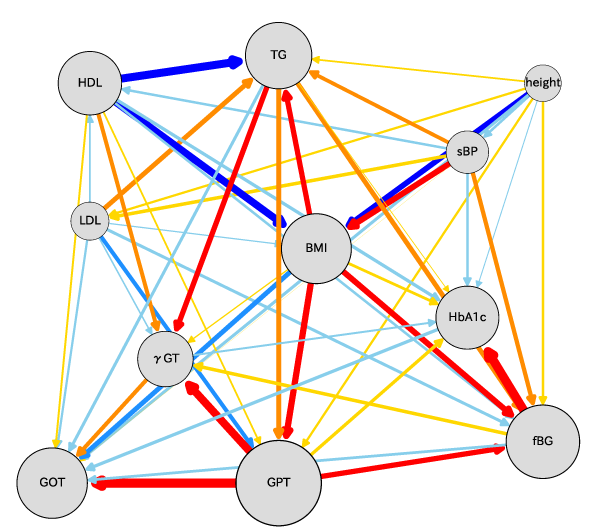
表6は推定された因果的順序のとおりに並んだ変数を行名と列名にとり,列方向の変数を原因,行方向の変数を結果として回帰モデルを作成した場合の偏回帰係数である.ここでも因果的順序の推定時と同様に,最も出現数の多かった因果的順序をもとにブートストラップ法を用いて係数行列を1,000回推定し,その中央値を入力した.
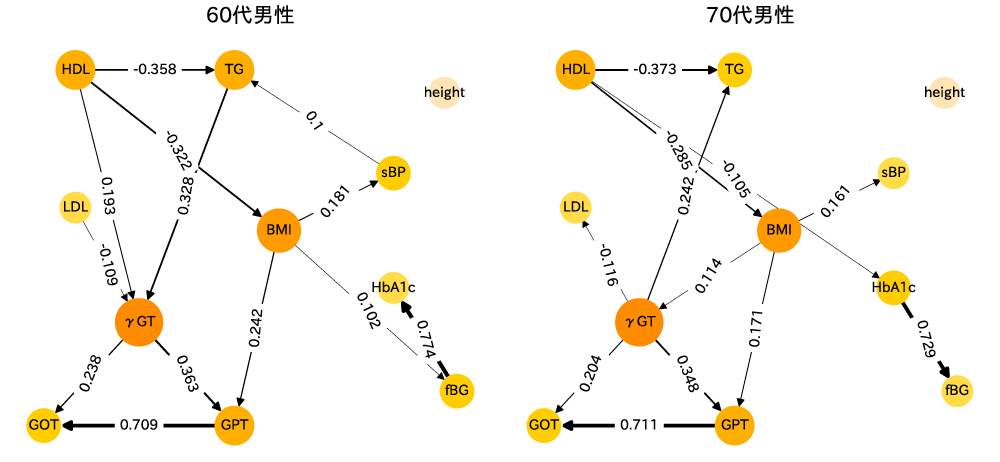
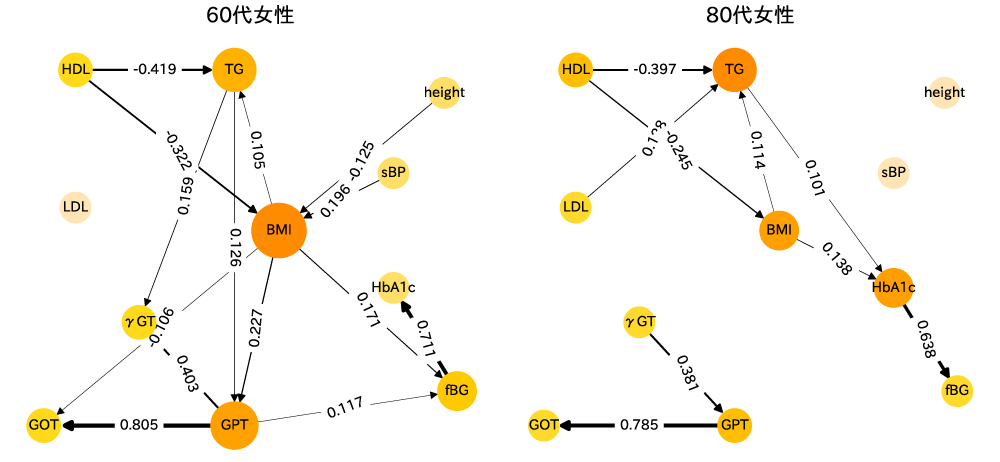
図10がLiNGAMモデルによって最終的に得られた因果ダイアグラムである.図中で,各項目同士が矢印で結ばれているところは,矢印の根本(原因)から先端(結果)に因果関係が推定されたことを示している.原因の変数が1標準偏差変化すると,結果の変数が何標準偏差変化するかを矢印の太さと色で示している(暖色は正の寄与,寒色は負の寄与を表し,矢印の太さは因果効果の大きさを表す).たとえば,HDLが増加すれば,BMIや中性脂肪,血糖値を改善(低下)させ,一方でBMIが増加することで,血糖値や肝機能の指標に悪影響をおよぼす(増加させる)ことを示している.同様に解析対象人数が30,000人を超えていた60代・70代男性の検診データから推定された因果ダイアグラム(図11)と,60代・80代女性の健診データから推定された因果ダイアグラム(図12)を示す.多少の違いはあれど,多くの因果関係が一致していることが見てとれるだろう.
7.これまでの総括と今後の展望
本研究ではDirectLiNGAMと呼ばれる因果関係を自動的に構築するアルゴリズムを用いることで,大阪府が持つ膨大な量の健診データから,生活習慣病因子間に存在する因果関係を明らかにした.本研究成果により,これまで経験に基づいた保健指導において,AIに基づいた解析を行うことによって保健指導による健康指標改善が具体性を持って可視化することが可能となった.これにより,保健指導の実施がエビデンスによって支えられ,指導を受ける側にも説得力を持って受け入れられやすくなるというメリットが期待できる.
LiNGAMモデルを用いることで,医学的な事前知識などに頼らずとも,原因と結果の関係を推定することが可能となった.一方,今回の結果から推定された因果効果と,これまでの医学的見地が一致していない関係や,エビデンスが不十分な因果関係を示すこともあった.たとえば,本解析ではHDLがBMIや中性脂肪に強い因果効果をもたらすことが明らかとなったが,医学の立場からこれらの因果関係を裏付ける十分なエビデンスが得られていないというのが実情である.このような結果が得られた理由としては,これらの変数は因果的順序で隣り合った近い順番で並んでおり,両者の因果的順序を決める指標に大きな差がなかったことや,本解析で取り入れることのできなかった潜在的な交絡因子の影響が観測できていなかったなどの理由が考えられる.たとえば,運動や食事はHDLとBMIの両方の変数に影響をおよぼす可能性があり,これらの変数を取り入れることができなかったため擬似的な因果関係が観測された可能性を否定できない.本研究で用いたLiNGAMモデルは非ガウス分布を仮定した連続分布のみを対象としているため,本解析に運動習慣や食習慣といった項目を含めることができなかった.今後は投薬や治療介入の有無,あるいは各疾患の有無といった離散変数まで取り込むようなモデルの開発を目指す.また,今日では傾向スコアなどを用いることで擬似ランダム化を仮定した因果効果の推定も盛んに行われており,本解析結果との比較検討にも着手したい.
参考文献
- 1)Kotoku, J., Oyama, A., Kitazumi, K., Toki, H., Haga, A., Yamamoto, R., Shinzawa, M., Yamakawa, M., Fukui, S., Yamamoto, K. and Moriyama, T. : Causal Relations of Health Indices Inferred Statistically Using the DirectLiNGAM algorithm from Big Data of Osaka Prefecture Health Checkups. PLoS ONE, Vol.15, No.12, pp.1-19 (Dec. 2021).
- 2)Shimizu, S., Hoyer, P. O., Hyvärinen, A. and Kerminen, A. : A Linear Non-gaussian Acyclic Model for Causal Discovery, Journal of Machine Learning Research, Vol.7, No.72, pp.2003-2030 (2006).
- 3)Shimizu, S., Inazumi, T., Sogawa, Y., Hyvärinen, A., Kawahara, Y., Washio, T., Hoyer, P. O. and Bollen, K. : DirectLiNGAM : A Direct Method for Learning a Linear Non-gaussian Structural Equation Model, Journal of Machine Learning Research, Vol.12, No.33, pp.1225-1248 (2011).
- 4)Thamvitayakul, K., Shimizu, S., Ueno, T., Washio, T. and Tashiro, T. : Bootstrap Confidence Intervals in DirectLiNGAM, In Proceedings ‐ 12th IEEE International Conference on Data Mining Workshops, ICDMW 2012, pp.659‐668 (2012).
- 5)Hyvärinen, A. and Smith, S. M. : Pairwise Likelihood ratios for Estimation of Non‐gaussian Structural Equation Models, Journal of Machine Learning Research, Vol.14, No.1, pp.111-152 (Jan. 2013).

大山飛鳥(非会員)ooyama@hacc.osaka-u.ac.jp
2021年帝京大学大学院医療技術学研究科診療放射線学専攻博士課程修了.博士(診療放射線学).現在,大阪大学キャンパスライフ健康支援・相談センター特任助教.機械学習技術を用いた生活習慣病等の疾病予測モデルの開発研究に従事.医学物理学会会員.

古徳純一(非会員)kotoku@med.teikyo-u.ac.jp
2004年東京大学大学院理学系研究科物理学専攻博士課程修了.博士(理学).現在,帝京大学大学院医療技術学研究科教授.大阪大学キャンパスライフ健康支援・相談センター招へい教授.第110回,第115回日本医学物理学会大会長賞受賞.数理的手法を武器とした医用システム開発の研究に従事.IEEE,AAPM,医学物理学会,日本物理学会,各会員.

土岐 博(非会員)toki@rcnp.osaka-u.ac.jp
1974年大阪大学大学院理学研究科物理学専攻博士課程修了.博士(理学).2010年大阪大学教授を退職.現在,大阪大学名誉教授・大阪大学キャンパスライフ健康支援・相談センター保健管理部門特任教授.専門分野は原子核理論物理,電磁ノイズの理論研究,健康に関するビッグデータの理論解析.日本物理学会論文賞受賞(1993,2011年).ドイツフンボルト賞受賞(2001).日本物理学会会員.
採録日:2022年10月26日
編集担当:澤邉知子(日本大学)