統合データベースプロジェクトから学ぶこと
1.生命科学データの共有と統合に向けて
生命科学のデータは膨大であるだけでなく種類も多く,また,計測時の文脈依存性などもあり,一言でいえば扱いにくい性質を多々備えている.そのため,単にそれらをデータベースの形で格納するだけでは十分に活用することができない.一方で,データの持つ潜在的な価値ゆえにデータは生産者に囲い込まれがちになる.そこで,15年ほど前より,文科省を中心に,生命科学分野の,公的資金により産出されたデータの共有を促進するとともに,データのフォーマットや意味の標準化を図り,複数のデータベースにまたがる解析を可能することにより(統合化),データ活用の利便性を大幅に高めるためのプロジェクト「統合データベースプロジェクト」が開始された.また,このプロジェクトの推進のためのセンタも整備された.この15年の間に,生命科学分野では,当初の想定を上回る形でデータの種類も量も増え,統合データベースプロジェクトの意義,アプローチも変化した.また,これを推進する国の体制,データ共有のルール,研究の進め方などにも大きな変化があった.もちろん世界的にも大きな動きが種々あった.本稿では,生命科学分野の統合データベースプロジェクトを取り巻く状況とその変遷を紹介することを通して,プロジェクトの成果と課題を総括する.内容は生命科学に特化したものではあるが,そこから得られる教訓はほかの分野のデーベースの開発・運用にも通用する面があるのではないかと考え,ここに紹介する次第である.
2.プロジェクト前夜 ―情報時代の幕開け―
これは大学共同利用研究機関法人の情報・システム機構(以下ROISと略す)の機構長をされていた堀田凱樹先生に教えていただいた話であるが,「生命科学ではおおよそ50年の周期で革命的なことが起きている.第1が1900年のメンデルの法則の再発見,その次が1953年のDNAの相補的な二重らせん構造の発見,そして第3が2001年のヒトゲノムの解読(正確には2001年はヒトゲノム概要配列の決定で,配列の完全決定は2003年)である」と.
最初の2つの発見の意義はさておき,3番目の2001年のヒトゲノムの解読は確かに生命科学に非常に大きな変化をもたらした.仮説を立てそれにかかわりそうなデータを実験で集め,検証するのではなく,最初にすべてのデータを網羅的に集めてしまって,それをベースに研究を展開するというアプローチが主流になった.今日データ駆動型科学と呼ばれる研究アプローチへの転換である.別の言い方をすれば生命科学研究において情報時代の幕が切って落とされたのである.ヒトゲノムの解読を受け,ほかの生物種のゲノム解読が促進されただけでなく,ゲノムの意味,機能を明らかにするために,ポストゲノムと呼ばれるプロジェクト,たとえば,すべてのタンパク質(プロテオームと呼ぶ,詳しくはコラム参照)の構造や機能を明らかにしようとするプロジェクト,なども続々と名乗りを上げた.
(コラム)オーム,オーミクス
genome(ゲノム)という単語の後半の-omeは「総体」を意味する接尾辞である.遺伝子すべてを表すgenome(gene+ome)に倣って,タンパク質すべてをproteome(protein+ome),mRNAなどの転写物全体をtranspriptome(transcripts+ome)などと呼ぶ.生体にはさまざまなオーム(ome)があり,ポストゲノム時代は多くの種類のオームが研究されるようになった.このような網羅的なデータの研究をオーミクス研究(omics)と呼ぶ.
ヒトゲノム解読の時期は,ちょうど20世紀から21世紀への変わり目と一致したため,我が国でもいわゆるミレニアムプロジェクトという形で,生命科学分野でも多くのオーミクスプロジェクトが実施された.誤解を恐れずに言えば,これらはすべて,膨大なデータを出すことを目的の1つとするものであった.ゲノムにとどまらずポストゲノムの観点からも,情報時代が大きく幕を開けたのである.
このような大規模データ生産の機運の高まりを受け,科学技術振興機構(以下JSTと略す)に2001年にバイオインフォマティクス(コラム参照)推進センター(以下BIRDと略す)が設置された.当時の科学技術庁に設けられたゲノム科学委員会の報告[1]を受けてのものであった.BIRDでは,ファンディングの形で「基盤的データベースの構築支援,情報時代に対応する研究開発,バイオインフォ人材育成」の3本柱が推進された.研究開発の公募では情報系と生物系の融合を目指す提案が求められた.データベース構築支援では,国際的なタンパク質立体構造データベースの日本拠点である日本蛋白質構造データバンク(以下PDBjと略す)や2018年にクラリベイト・アナリティクス引用栄誉賞を受賞したKEGGデータベースなどの発展の基盤がこの時に作られた.人材的にも,現在世界的に活躍している多くのバイオインフォマティクス研究者がBIRDの支援で育った.結局BIRDは2011年までの10年続いた[2].
(コラム)バイオインフォマティクス
この学問分野は,その名の通り,バイオ(生物)のためのインフォマティクスを研究する分野であるが,その後学問が進展して,インフォマティクスを使ってバイオを研究する分野の意味でも使われるようになった.また,バイオの意味も基礎生物学だけでなく,医学や農学などの応用科学の意味も含有するように変わってきている.なお,BIRDが設立された頃は,バイオインフォマティクスという言葉はそれほど普及していなくて,BIRD設立のきっかけとなった政府の報告書ではゲノム情報科学と呼ばれていた.
ところで,情報時代の幕開けは,実は,BIRD設立の10年前の1990年頃にはすでに予見されていた.1990年は,世界的にヒトゲノムプロジェクトが走り出した時期に当たるが,その当時の米国の計画[3]には,データベースや情報解析がこのプロジェクトの成否を握ると書かれている.このような認識の下,我が国でもヒトゲノムプロジェクトの推進センターとして1991年に東京大学医科学研究所に設置されたヒトゲノム解析センターでは当初設置の3講座のうち2講座は情報系の講座―データベース分野とDNA情報解析分野(どちらも当時の名称)―であった.個人的なことで恐縮であるが,筆者は縁あって当時在職していた九州大学から設置まもないヒトゲノム解析センターデータベース分野に移りデータベース開発を担当することになった.これを契機として今日まで生命科学系のデータベース開発に携わることになった.
3.統合データベースプロジェクトいよいよ始まる
2000年頃に幕を開けた情報時代であったが,その後の展開は質,量ともに予想をはるかに超えるものであった.2000年頃はゲノム研究もポストゲノム研究も基礎研究としての意味合いが強かったが,2005年頃に出現した次世代シークエンサー(以下NGSと略す)と呼ばれるゲノム配列決定装置の革命的とも言える進歩が世の中を大きく変えた.30億塩基からなるヒトゲノム1人分相当を決めるのに,2000年頃は100億円規模の予算が必要であったが,その後10年ほどの間に10万円ほどまでにコストが下がり,医学などへの応用が一気に花開くことになった.NGSの性能の伸びはムーアの法則をはるかにしのぎ10年ほどで性能が1万倍になった.これにより生命科学がまさにビッグデータ分野の1つと認識されるようになったのである.
もう1つの展開は,先に述べたポストゲノムプロジェクトの動きである.NGSほどではないが,質量分析器や顕微鏡などにも大きな技術革新があり,ゲノム以外でも精度の高いデータがより安価に大量に得られるようになった.これによりそれらのデータを格納したデータベースの重要性が飛躍的に高まり,多くの研究者が独自のデータベースを自前で作り情報を発信するようになった.
その一方で,前述のミレニアムプロジェクトなどで作られた多くのデータベースがプロジェクトの終了と同時に維持管理の費用が捻出できずに捨てられる,あるいは,どこかに死蔵されるという問題も出てきた.プロジェクトの多くはデータ生産が大きな目的の1つであったにもかかわらずこのようなことが起きてしまったのである.BIRDで支援されていたものは基盤的なデータベースに限られていたため,残念ながら,それ以外のデータベースはこのような運命を辿ることになったのである.
このようなデータやデータベースをめぐる状況を背景にして,内閣府を中心に2005年頃より統合データベースプロジェクト(以下,統合プロジェクトあるいは単にプロジェクトと略す)の構想が浮上したのである.その当時内閣府で連携施策群「生命科学の基礎・基盤」の推進という施策が行われた.これは各省庁で実施されている施策に重複がないか,あるいは,欠落がないかを問うものであった.この中で統合データベースの必要性が浮上したのである.背景には,先ほど述べたように,生命科学分野で大量にデータが産出されているが,それらが死蔵され,あるいは,バラバラにデータベース化され,有効活用されていない(統合データベースの欠如)という状況があった.科学技術振興調整費を使って国内外のデータベースの構築状況とその背後にある問題が調査された[4].国立遺伝学研究所の大久保公策教授を中心とした調査チーム[5]により,我が国において「データ共有のルール作り」と「その受け皿となるセンター」の必要性が明らかになった.
内閣府の調査結果を受ける形で,文科省,経産省,農水省,厚労省それぞれにおいて,相次いで,統合プロジェクトあるいはそれに類するプロジェクトが立ち上がった.以下の章では,これらの中で一番規模が大きく,また,永続的に進められてきた文科省のプロジェクトを中心に紹介することとする.
4.諸外国におけるデータベース構築とデータベースセンタ
ここで,生命科学分野のデータベースに関して,外国での動きにも少し触れておこう.
米国では,1964年に文献データベースPubMedの前身が,1969年に遺伝子変異疾患データベースMIMが,1971年にタンパク質立体構造データベースPDBが,1982年に核酸塩基配列データベースGenBankが,それぞれ立ち上がっている.欧州では,1980年に核酸塩基配列データベースの前身が作られている.これらは現在,生命研究になくてはならないデータベースになっている.
ところで,これらのデータベースが現在も更新され,有効に機能している背景には,
- (A)データの共有(データベースへの登録)が義務化されていること,
- (B)その受け皿である恒久的センタが存在すること,
の2点が挙げられる.前者に関して言えば,公的資金配分機関(米国だと国立衛生研究所(以下NIHと略す)によるものと出版社によるものとがある.多くの出版社は論文投稿前にそれの根拠となるデータをデータベースに登録することを義務化している.これらのデータ共有の圧力により,常に最新のデータが漏れなくデータベースに入っているのである(注:生命科学のすべての種類のデータについて出版前のデータ登録が義務化されているわけでない.しかし,その種類はどんどん増えている)
なお,ヒトゲノムプロジェクトでは,データの即時(解析後24時間以内の)公開と自由な利用に関して関係者の間で1996年に合意が形成された.これをバミューダ原則と呼ぶが,このような考えが生命科学にはしっかりとしてあり,データの共有が進む大きな要因となっている[6].
さて,上記(B)で言及したデータベースセンタとしては,米国のNCBIと欧州のEBIがある.これらはそれぞれ1988年,1992年の創立である.これらのセンタの規模感であるが,数十億円から百億円の予算規模で,数百人規模の研究員が雇用され,数十ペタバイトの容量のディスクが設置されている.なお,日本では1987年に国立遺伝学研究所にDDBJセンターが設置され,NCBI,EBIと連携して国際塩基配列データベースを協同運営している.このデータベースは世界中の誰でもデータの登録や利用ができるもので,人類の共有財産となっている.
データ共有,データベース構築については,最近,国内外でさまざまな動きがあり,それについては後ほど改めて触れることにする.データベースの歴史,統合プロジェクト初期の取り組みなどについて興味のある方は,少し古いもので恐縮だが,文献[7]を参照されたい.
5.DBCLS時代 ―統合プロジェクト第1期―
先に述べたように,内閣府の動きに呼応する形で,2005年に文科省ライフサイエンス委員会の下にデータベース整備戦略作業部会が作られ,その報告[8]を受けて,2006年後半より統合プロジェクトが立ち上がった.このプロジェクトは2006年9月から2011年3月までの4年半の時限的プロジェクトとしてスタートした.正確には2006年は半年間の試行的プロジェクトとして,2007年4月から4年間の本格プロジェクトとして実施された.そして,2007年4月には,このプロジェクトの推進センタとしてROISにライフサイエンス統合データベースセンターDBCLSが設立された.
ROISにはその配下の国立遺伝学研究所にDDBJセンターがすでに存在していたが,種々の事情から,同じROIS内にもう1つのセンタDBCLSが作られることになった.これが後々に尾を引くことになる.このことについては最後に触れる.
さて,統合プロジェクト設立に至る,我が国の生命科学分野のデータベースの置かれた状況を図1に示す.これはその当時私が統合プロジェクト説明のために使用していたスライドである.また,図2にその当時のプロジェクトの考え方を示す.この解説は割愛するが,当時(約15年前)の状況がそれなりにご理解いただけるものと思う.


ところで,この期に及んで恐縮だが,なぜデータベースの統合が必要なのだろうか? それは生命科学のデータには厄介な性質があるからである(図3).生命科学以外でも類似の性質を持った分野があるのかもしれないが,ここでは生命科学特有と表現しておく.生命研究から出てくるデータはこのような性質を持っているので,そのままではほかの研究者が出したデータをうまく活用できない.そこで,データやデータベースの統合(ID,専門用語などオントロジーやフォーマットを揃える)が必要になるのである.生命科学は目的や実験手法が異なる多数のプロジェクトから成り立っているが,それらのプロジェクトごとに遺伝子などの実体は同じなのに違うIDがついていたりしていて,また,データ取得時の文脈依存性などがあり,それらを寄せ集めて「ビッグなデータ」としても正しい統計処理や機械学習が行えない.生命科学のデータはそのままでは新たな仮説や規則を生み出す基盤という意味でのビッグデータ足りえず,それを統合して初めてビッグデータとして価値のあるものになるのである.統合プロジェクトはそのためのものである.もちろん,個々人のゲノム情報などは個人情報保護の対象なので,統合化処理に加え,匿名化やデータへのアクセスに制限を課すような運用も必要になる.

ここで,DBCLS時代の統合プロジェクトの主な成果と問題点を挙げておく.第1の成果は,公的資金で行われた研究から得られたデータをデータベースに登録することのお願いを協力依頼という形でその研究の公募要領に記載してもらったことである(科学研究費の記載例を図4に示す).協力依頼の文言は,最初は文科省ライフサイエンス課の委託プロジェクトに記載され,その後,科学研究費や他省庁などの研究公募にも広がっていった.データ共有の義務化までにはなっていないが大きな前進であった.

成果の2つ目は,統合プロジェクトが,文科省,経産省,農水省,厚労省,の4省連携で実施されたことである.これはその後2011年の4省合同ポータルサイトに結実することになった.細かいことは省略するが,今でもこの連携体制は基本的に維持されている.4省の連携に際しては,最初に統合の道筋に関して合意がなされ,それに沿ってプロジェクトが進められている.そのロードマップを図5に示す.現在は第4段階の「再構築」のフェーズにある.

成果の3つ目は,多様化・複雑化するデータの標準化に関してデータベース開発の実務者による国際連携の仕組みが日本主導でできたことである.生命分野のデータ産出において,日本からの貢献は1割もない(DDBJセンターへのデータ登録で見るとおおよそ数パーセント)と推測される.このような状況では,外国で産出された膨大なデータと組み合わせて活用することが不可欠であり,その点で,フォーマット,オントロジー,IDなどが日本固有のガラパゴス的なものでは意味がない.
この問題を解消するために,DBCLSでは2008年より毎年国際バイオハッカソン[9]を開催してきた(2011年より後述のバイオサイエンスデータベースセンター(以下NBDCと略す)も主催者に加わることになった.2020年はコロナ禍のため国際版は開催中止).外国のデータベース開発実務者を毎年日本に招いて1週間の合宿を行い,データの標準化や連携を図ってきた.ハッカソンの成果は2019年に運用を開始したbiohackrxivプレプリントサーバ(https://biohackrxiv.org/)で公開されている.各国のデータベース開発者との議論の中から多くの成果が得られたが,その1つがオープンサイエンスにおけるデータ公開の適切な実施方法を表現したFAIR原則(コラム)誕生への貢献である[10].
(コラム)FAIR原則
Findable, Accessible, Interoperable, Reusableの頭文字を取って命名された原則.データの共有,公開の際に守るべき規範を示したもの.これを守ったデータはオープンサイエンスで利活用しやすい.興味のある方は以下の解説記事(英文の日本語訳)や本特集号の解説論文[11]を読んでいただきたい.
https://biosciencedbc.jp/about-us/report/fair-data-principle/
さて,DBCLS時代の問題は,統合プロジェクトが多くの機関で分担して行われたことである.図6にその当時の体制図を示す.多くの機関,多くのデータベースがオールジャパン的に参加・連携したと言う点では良かったが,分担機関の選定にDBCLSが直接関与してなかったこともあり,各データベースのバラバラ感は残ってしまった.この当時は連携構築やデータ共有の仕組み作りの問題への対応が大変で,各データベースで使用するIDの統一などまでは十分に踏み込めていなかった.それは次のNBDC時代まで持ち越された.
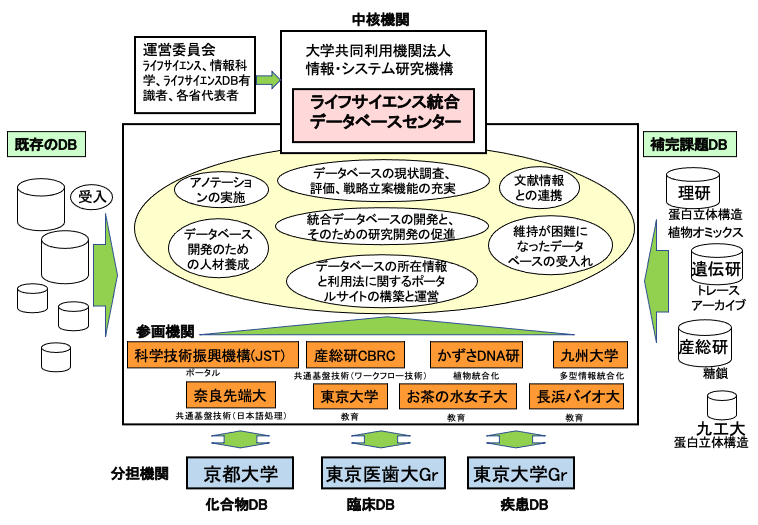
DBCLSの活動の成果の詳細について興味のある方は,下記のサイトを参照いただきたい.
6.NBDC時代 ―統合プロジェクト第2期―
さて,前章で述べたように,文科省の統合プロジェクトはまずは4年半の時限プロジェクトとして実施された.当然のことながら4年半後にどうするかということが問題となった.データベースは永久にとは言わないまでも,ほんの数年で運用をやめてよいものではない.2011年3月の時限プロジェクト終了に向けて,当時の内閣府総合科学技術会議および文科省それぞれでこの問題を議論する委員会が作られ,それぞれ報告書が取りまとめられた[12],[13].その結果,先行していたBIRDの役割や機能もある程度引き継ぐ形でJSTにNBDCが作られることになった.ただし,DBCLSは残し,NBDCでは困難な統合技術開発と人材育成機能を担うことになった.NBDCでは,戦略機能,ポータルサイト運営機能,ファンディングによる分野ごと(たとえば,植物分野とか微生物分野など)の統合データベース構築を担うこととなった.
NBDC(+DBCLS)の活動の主な成果と問題点を以下に述べる.成果としては,日本人ヒトゲノムデータベース構築[14]とその2次データベース であるTogoVar[15]開発がまず挙げられる.前者は主に日本人ゲノムの変異をアーカイブしたデータベースである.米国のdbGap,欧州のEGAに相当するものである.TogoVarは,外国で作られているヒトゲノム変異データベースや関連文献と,日本人ゲノム変異のデータ(NBDCヒトデータベースからの分も含め)とをまとめてワンストップで検索可能にしたシステムである.NBDCヒトデータベースもTogoVarもどちらも今では我が国のゲノム医科学研究に欠かせないデータベースとなっている.
ところで,ヒトゲノムは究極の個人情報であり,その運用には「人を対象とする生命科学・医学系研究に関する倫理指針」などの指針を遵守することが求められる.上記ヒトゲノムデータベースを運用するにあたり,指針を踏まえたデータ共有ガイドラインや情報セキュリティガイドラインが必要となる.このようなガイドライン[14]を作成したことも本プロジェクトの大きな成果だと考えている.このようなガイドラインは本来は国主導で決めることが望ましいが,種々の事情からそれが叶わなかったので,NBDCが率先して策定したものである.これらのガイドラインは,ヒトゲノムなどの機微データを扱うほかのデータベース(日本医療研究開発機構(以下AMEDと略す)のAGDなど)で参考にされるまでになっている.
NBDCのもう1つの成果はRDF(Resource Description Framework)によるデータ統合である.図3に示したように,生命科学データは曖昧性,文脈依存性,などの特徴があり,それゆえ,データベースごとにIDやオントロジーがバラバラである.この辺りの整理をしないと,複数のデータベースにまたがる検索や知識発見は簡単には行えない.我々はRDFを用いて,主要なデータベースを記載しており,これにより複雑で柔軟な検索を可能としている.もちろん,ゲノム配列や画像などはRDFには馴染まないので,それはそれでRDF以外の枠組みでデータ統合を図っている.
NBDCの活動の成果の詳細について興味のある方は,下記のサイトを参照いただきたい.
なお,このサイトには統合プロジェクトの目標の解説や各種調査結果なども記載しているので,ご覧いただければ幸いである.たとえば,細胞工学という雑誌に2011年から2012年にかけて掲載された連載「我が国のデータベース構築・統合戦略」の原稿なども下記から見ることができる.
https://biosciencedbc.jp/about-us/report/
7.目まぐるしい動き
NBDCができてちょうど10年になるが,この間,当初の想定を超えるような動きが国内外でさまざま起きている.生命科学やバイオ産業を取り巻く状況は激しく変化している.とてもすべてを紹介する紙面はないが,皆さまの関心ありそうなものを少しだけピックアップして紹介する.
7.1 さらなるビッグデータ
第2章,第3章で述べたビッグデータ化の動きはさらに加速・拡大しつつある.医学分野では,世界各国で数十万人規模でゲノムその他のオミックスデータ,臨床情報,生活習慣情報を(時系列にそって)網羅的に集めるプロジェクトが進行中である[16].また,診療録レセプト,疾患レジストリ,などのいわゆるリアルワールドデータの活用も視野に入ってきた[17].また,このようなデジタルトランスフォーメーション(DX)化の動きは,医学に限らず,農業分野などでも起きてきており(例:バイオエコノミー[18]),生命研究,バイオ産業が新たなステージに入ろうとしている.さらに,これからは,オープンなデータだけなく,民間企業などが保有するクローズドなデータとを組み合わせた統合解析,などの新しい技術も必要になってきている.国の方でも健康・医療戦略[19],バイオ戦略[20]を立て積極的な展開を始めている.
7.2 国際連携の広がり
医学分野では,GA4GH[21]という国際アライアンスがヒトゲノム情報などの共有に関して積極的な活動を展開している.さまざまな取り組みがあるが,たとえば,機械可読なインフォームドコンセント(これによりこれとユーザ認証を組み合わせることによりどのデータは誰に見せて良いか機械的に判断できる),動的インフォームドコンセント,データビジティング(機微データをほかのサイトに持ち出さずに,機微データのある場所に解析プログラムを送り込んで解析する方法),など新しい仕組みの提案がなされている.一方で,オープンサイエンスに逆行する動きもある.生物多様性条約の名古屋議定書にある「遺伝子資源の利用から生ずる利益の公正かつ衡平な配分を実現する」のルールを遺伝子配列などのディジタル配列情報にも拡張する動きがそれである[22].今後注意が必要である.
8.統合プロジェクトから学ぶこと ―成功に導く10の教え―
これまで約15年(BIRD時代も入れると20年ほど)にわたって,我が国の生命科学分野のデータベース整備に携わってきた.それなりの苦労があったがどこまで一般化できるものかは分からない.また,前述したように,生命科学のデータはほかの分野とは違う性質を持っている.このような事情から,どこまで読者に関心のある内容になるか自信がないが,私がこれまで経験したこと,学んだことを以下,10のポイントにまとめ,述べることとする.もし,ほかの分野でのデータベースセンタ設置やデータベースプロジェクト遂行の参考になれば,この上ない喜びである.
8.1 統合センタの「統合」を
データベースの統合を目的としてDBCLSやNBDCが設立されたが,これ以外にもこの分野ではDDBJや阪大のPDBjなどもあり,これらの連携をどう図るのか難しい問題である.私はDBCLS,DDBJ,NBDCのそれぞれのセンタ長経験者と言うことで,これらの連携に努力してきたが,個人できることには限界もある.これらのセンタは設立時にそれなりのロジックがあって設置されたわけであるが,現在はそのセンタが所属する親組織のロジックもあり一体的な運用は容易ではない.これらのセンタはすべて文科省系のセンタであるが,農水省,厚労省,経産省,さらにAMEDにもそれぞれデータベース(センタ)のアクティビティがあり,これらを我が国としてどう連携,連動させるかは今後の大きな課題である.
「教訓1:データの統合には,センタの統合を」
8.2 まずはデータ共有ポリシー策定を
生命科学はほかの分野に比べてデータ共有が比較的進んでいると言われている.これは先に述べた欧米の公的資金配分機関と出版社の方針によるところが大きい.我が国でも,第5章で述べたように科研費などの公募要領にデータ提供の協力依頼の記載があり,AMEDなどでもデータ共有のポリシーが定められてきている.AMEDの末松前理事長が,No Share, No Fundと発言されたこともある.しかしながら,欧米のそれに比べると共有の強制力という点ではまだまだ弱いと言わざるを得ない.これがしっかりしないと受け皿としてのセンタを作っても機能しない.DBCLS,NBDCの苦労の多くもこの点に関するものが大きかった.今後の改善が望まれる.
「教訓2:データの受け皿作りの前に,データ共有ポリシーを確立すべし」
8.3 データ提供者を高く評価せよ
強力なデータ共有ポリシーは我が国にぜひとも必要だが,義務化だけでは不十分である.研究者の理解と協力を得るには,データ提供した際のインセンティブ付与やデータ登録の支援も重要である.現在の論文至上主義から転換し,他人に有用なデータを提供することに大きな価値を見出し,それを評価する仕組みが必要である.ビッグデータは宝の山と言われる.それならば,その宝を提供した研究者を評価すべきである.このことは研究の文化ともかかわる大きな問題だが,これを進めないとデータ駆動型科学実現への道は遠い.
「教訓3:多くの良いデータを提供した研究者を評価する文化を醸成すべし」
8.4 最初が肝心
我が国では,上に書いたように,データ共有ポリシーがなかったり,弱かったりする.それゆえ,データ産出プロジェクト立案時のDMP(データマネージメントプラン)の記載がいい加減だったり,DMPの遂行状況がプロジェクトの評価に反映されなかったりという問題がある.そのため,データベース作りもいい加減になりがちであるし,その後の利活用のこともあまり意識されなかったりする.これまでのデータベース統合は,プロジェクトが終了してから行われていたが,これでは散らかったデータを整理するのにコストがかかりすぎる.プロジェクトの企画立案段階からNBDCなどのデータベースセンタと一緒にデータベース作りを始める必要がある.
「教訓4:最初からデータベースの統合,利活用を意識したプロジェクト立案と実行を」
8.5 パーマネントポジションの確保を
データベースの開発,更新,維持管理には専門人材が欠かせない.とくに生命科学分野では,データの整理統合のためには生物学医学の知識だけでなく,生命倫理,個人情報保護などの知識も必要となる.もちろん,データベース,スパコン,情報セキュリティ,などの情報系の知識,さらには計測技術の知識も欠かせない.しかしながら,先に述べたDBCLS NBDC DDBJなどは雇用人数が少ない上に,ほとんどが任期付きである.欧米のセンタでは第4章に書いたように数百名規模の専門人材がいる.このような状況では高度で安定的なデータベースの運用ができない上に,欧米と伍して行くのは非常に難しい.我が国では,国を挙げてSociety5.0,DX,データ駆動型科学・社会を推進と言う割には,この点が大変お粗末である.至急改善が必要で,そうしないと,そして誰もいなくなったになりかねない.
「教訓5:データ駆動型の推進にはパーマネントポジション用意せよ」
8.6 データベースの価値を測れ
一般にデータベースの開発,運用には大きな費用がかかる.しかしながら,この費用に見合った,あるいは,それ以上の価値があることを,財政当局に理解してもらうのは容易ではない.個々のデータベースに何万人,何十万人の利用者がいれば,理解を得るのはそれほど困難ではないかもしれないが,多様な生命科学研究では,一部のデータベース(たとえば,生物種横断的な基盤的なゲノムデータベースやタンパク質データベース)を除いて,そうなるまでには時間もかかるし,それほど利用者が多くなくても欠かせないデータベースもある.この問題を解消するには,客観的にデータベースの価値を測る指標を開発する必要がある.私には良いアイディアはないが,たとえば,第4章で紹介した欧州のEBIが出しているレポート(EBIの価値の見積もりを試みたもの)などは参考になるかもしれない[23].ぜひ読者の皆さんのお知恵を拝借したい.
「教訓6:データベースの価値を数値で示せ,そうでないとデータベースは維持できない」
8.7 再利用性の高いデータを探せ
第2章で述べたように,ゲノムプロジェクトの成功以来,各国でオームデータ産出プロジェクトが多数立案され,実行されてきた.世界中ではその数は数千とも言われる[24].しかしながら,これらのすべてのデータについてデータベースを開発し,それを運用するのは費用的に無理である.この中でどの種類のオームが多くの研究者にとって将来にわたり再利用性が高いのかを見極める必要がある.これも研究の進展とともに変化する面もあり,容易ではないが,これをしないと無限の予算が必要となり,財政当局の理解は得られない.
「教訓7:ビッグデータの中から本物の宝を見つけ出せ」
8.8 新たなデータビジネスモデルの構築
DBCLS,NBDCではこれまでオープンなデータを対象としてそれを統合データベース化し,無償で利用者に提供してきた.このための予算はすべて文科省からいただいてきた.世界的にもほとんどのデータベースは無償で利用できるようになっている.しかし,今後の持続性を考えると,一部有償化する,データ産出プロジェクトから消費税方式で運営費を集める,などの方式も検討すべきであろう.この点もお知恵を拝借したい.なお,持続的な生命科学データ基盤の構築の議論に関しては日本学術会議の提言が詳しい[25].参考にされたい.
「教訓8:持続性確保には新たな資金獲得の道も探れ」
8.9 分野の特性を考慮せよ
繰り返しになるが,生命科学データはほかの研究分野やビジネスのデータに比べて厄介な性質を持っている.そのため,機関別ではなく,データの特性に応じた分野別のレポジトリを作ってデータの整理統合を図る必要がある.また,データの統合利用や利活用にはSociety5.0などで採用されているAPIを作るだけでは不十分である.そうしないとデータの有効活用は進まない.生命科学では,まさに統合プロジェクトが必要だったのである.
「教訓9:分野の特性を見きわめてデータベースやレポジトリを作れ」
8.10 自らデータ駆動科学の実践を
上にいろいろ書いたように,データベースの開発,運用の費用を持続的に捻出するためには,さまざまな取り組みが必要である.その中でも,最も重要なことはデータベースから素晴らしい成果が生まれることに尽きる.ノーベル賞受賞の山中伸弥先生は,体細胞の初期化にかかわる山中4因子はデータベースの助けがあってのことだと言ってくださるが,それは大変稀な例で,データベースがあったから,こういう研究ができたと誰かが言ってくれることはなかなかない.データベースの開発者自らがお手本を示すことも必要だ.実行するのは至難の業ではあるが.
「教訓10:データベースの成果は自分で作れ」
9.30年後の展望
人生の多くの時間をデータベース作りに費やしてきた.これにより我が国のデータ駆動型科学がこんなに進みましたと胸を張って言いたいところだが,とてもそこまでには至っていない.道半ばである.生命科学が50年周期で大きな変革が起きるとすれば,次は2050年頃だ.その変革にデータベースが不可欠な役割を担っていてほしいとは思うが,よく分からない.次の変革を自分の目で見ることは到底叶わないが,生きている間に少しはその片鱗を見たいものだ.
参考文献
- 1)科学技術会議ライフサイエンス部会ゲノム科学委員会:ゲノム情報科学におけるわが国の戦略について(平成12年11月)(要約),
https://www.lifescience.mext.go.jp/download/1th_database/1d-2.pdf - 2)バイオインフォマティクス推進センター:https://www.jst.go.jp/nbdc/bird/info.html
- 3)ヒトゲノム解析計画―遺伝情報を解読する巨大プロジェクトの全容 米国議会技術評価局(OTA)報告書:Newton special issue(日本語)(1990).
- 4)平成17年度科学技術振興調整費「科学技術連携施策群の効果的・効率的な推進」の審査経緯及び結果概要について:
https://warp.da.ndl.go.jp/info:ndljp/pid/286184/www.mext.go.jp/b_menu/houdou/17/10/05102801.htm - 5)生命科学データベース統合に関する調査研究:
https://warp.ndl.go.jp/info:ndljp/pid/286794/www.mext.go.jp/b_menu/houdou/17/10/05102801/001/001.pdf - 6)高祖歩美:生命科学分野におけるデータ共有のあゆみ,情報処理,Vol.54, No.12, pp.1226-1231 (2013).
- 7)ライフサイエンス統合データベースの推進方策について(報告):
https://www.rois.ac.jp/open/pdf/db_houkokusho.pdf - 8)我が国におけるライフサイエンス分野のデータベース整備戦略のあり方について:
https://www.lifescience.mext.go.jp/download/news/report_DB.pdf - 9)BioHackathon:http://www.biohackathon.org/, https://biosciencedbc.jp/event/biohackathon/
- 10)FAIR原則:https://biosciencedbc.jp/about-us/report/fair-principle/
- 11)青木学聡:オープンサイエンスと研究データ管理の動向,デジタルプラクティスコーナー,情報処理,Vol.62, No.5 (May 2021).
- 12)統合データベースタスフォース報告書:https://www8.cao.go.jp/cstp/project/bunyabetu2006/life/14kai/siryo1-2.pdf
- 13)ライフサイエンスデータベースの統合・維持・運用のあり方:
https://www.lifescience.mext.go.jp/files/pdf/n676_s1.pdf - 14)NBDCヒトデータベース:https://humandbs.biosciencedbc.jp/
- 15)TogoVar:https://togovar.biosciencedbc.jp/
- 16)諸外国におけるゲノム医療の制度・体制・運⽤等に関する調査(概要版):
https://www.kantei.go.jp/jp/singi/kenkouiryou/genome/genome_dai2/sankou3.pdf - 17)リアルワールドデータを活用する鍵:
https://www.mri.co.jp/knowledge/mreview/201907-5.html - 18)バイオ×デジタルによる新たな経済社会(バイオエコノミー)に向けて:
https://www.nedo.go.jp/content/100870410.pdf - 19)健康・医療戦略:https://www.soumu.go.jp/main_content/000691940.pdf
- 20)バイオ戦略2020:https://www8.cao.go.jp/cstp/bio/bio2020_honbun.pdf
- 21)GA4GH (Global Alliance for Genomics and Health) : https://www.ga4gh.org/
- 22)有田正規:経済化される生物多様性,科学,Vol.88, No.7 (2018).
- 23)The Value and Impact of the European Bioinformatics Institute : Full Report (Jan. 2016), https://beagrie.com/static/resource/EBI-impact-report.pdf
- 24)Big biology: The ’omes puzzle :
https://www.nature.com/news/big-biology-the-omes-puzzle-1.12484 - 25)日本学術会議提言:持続可能な生命科学のデータ基盤の整備に向けて,
http://www.scj.go.jp/ja/info/kohyo/pdf/kohyo-24-t279-1.pdf
高木利久(正会員)tt@tuins.ac.jp
東京大学工学部計数工学科卒業.九州大学,東京大学勤務を経て,現在富山国際大学学長科学技術振興機構バイオサイエンスデータベースセンター長兼務.これまで情報・システム研究機構ライフサイエンス統合データベースセンター長,国立遺伝学研究所DDBJセンター長を勤めた.
編集担当:藤原一毅(国立情報学研究所)