ライフサイエンス研究におけるクラウドソーシングの利用と実践
1.はじめに
近年,ライフサイエンス研究分野では,インターネット経由で群衆(クラウド)を活用した研究論文が増えている.海外では多数のクラウドワーカを活用した研究が行われているが,日本のライフサイエンス研究の現場では,活用事例やクラウドソーシングの利用情報が少ないために,研究応用への敷居が高いと考えられる.本稿では,ライフサイエンス研究分野で研究開発に従事する者が,クラウドソーシング応用研究を実施できるように,既報研究の分類や,クラウドワーカ活用プラットフォームの参考情報を紹介する.また,筆者らがライフサイエンス研究やDNAデータバンク事業で培ってきたクラウド活用の知見と課題を紹介する.さらに,注意点として,ライフサイエンス研究分野で群衆参加型研究を研究雑誌に投稿する際に必要となる,研究倫理審査承認について,具体的な手続き方法を説明する.
2.ライフサイエンス分野でのクラウドソーシング研究の分類
2.1 クラウドソーシング研究の歴史
群衆参加型研究がクラウドソーシング研究と呼ばれ始めたのは,Jeff Howeが造語「クラウド+ソーシング」を定義した2006年頃からといわれている[1].群衆参加型研究の歴史は古く,1714年に英国でコンテスト形式で船上で経度を計測する方法が募集された[2].群衆参加型の科学は,市民科学(シチズンサイエンス)研究と呼ばれている[3].ライフサイエンスの分野では,2002年開始の米Cornell大のeBirdプロジェクト[4]が代表的な市民科学プロジェクトとして知られている.eBirdは,市民が収集した鳥の緯度経度観測情報を用いて,研究者は時空間統計モデルによる生物種地理分布を推定する.また収集した鳥の写真7,000万枚を使い,400種の鳥の画像分類ソフトウェアを提供している.データ収集目的のクラウドソーシングプロジェクトでは,eBirdのような観察型の生物多様性研究(ダニコレクション[5]等)や,参加者を被験者とするヒト疾患研究(心血管疾患リスク[6], 食品媒介疾患:Iwaspoisoned.com[7]など)がある.
2.2 データライフサイクルによるクラウドソーシング・タスクの分類
群衆が参加するクラウドソーシング研究開発タスクを,データライフサイクル[8]の観点から本稿では3種類に分類する.研究起案や仮説生成の段階を除いて,①サンプリング, ②アノテーション, ③モデリングの3種類とする.サンプリングは,試料収集・試料センシングなどのデータ収集タスクを指す.生物試料を収集する以外に,参加者が被験者となり生体試料を提供する場合もある.アノテーションは,主にデジタルデータの編集を通してデータ構造化作業を行う.モデリングは,データをモデル化する.たとえば機械学習予測モデルを構築するタスクを指す.
2.3 参加の動機によるクラウドソーシング・タスクの分類
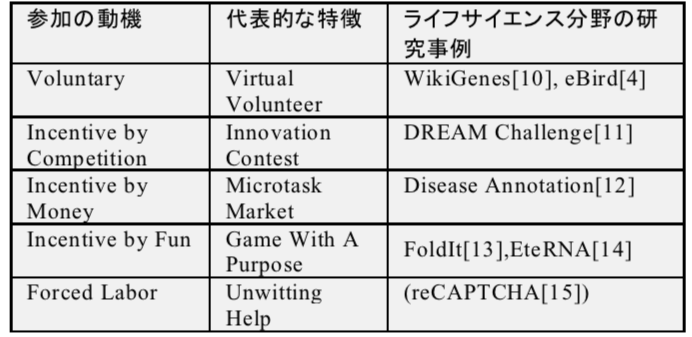
動機の観点でもクラウドソーシング・タスクの分類が報告されている[9].本稿では参加の動機を5クラス(ボランティア,競争,金銭報酬,面白さ,強制)に分類する. 表1にライフサイエンス分野の事例をまとめた.
動機がボランティアの場合は,代表的なライフサイエンス分野での研究事例はWikiGenesとeBirdである.WikiGenesは,遺伝子注釈情報を作業者名のログとともにWikiでデータベース化する.動機が金銭報酬の場合の代表的な特徴は,マイクロタスク仲介市場が挙げられる. Amazon Mechanical Turk (AMT)を使ったGoodらの疾患注釈などが事例である.ほかには,競争や張り合いが動機のイノベーションコンテストと,ゲームの楽しさが動機のGame With A Purpose (GWAP) がある.GWAPはゲームの副産物として,注釈作業が達成されるクラウドソーシングの仕組みを指す.各々のライフサイエンス分野の有名事例は,DREAM ChallengeとFoldItになる.reCAPTCHAは,Von Ahn博士が考案したクラウドソーシングの仕組みで,強制的な作業(Forced Labor)の裏で,意図せず(Unwitting)に文書の電子化等の有用な注釈データが蓄積される.
2.4 募集期間・参加者数・データ数の事例
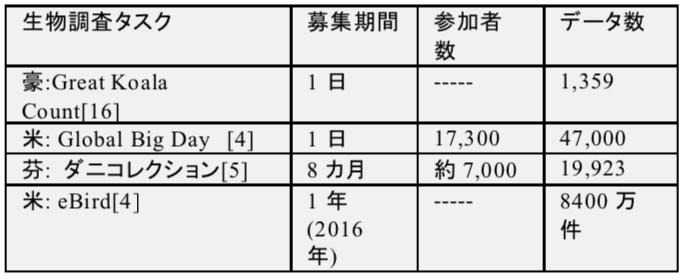
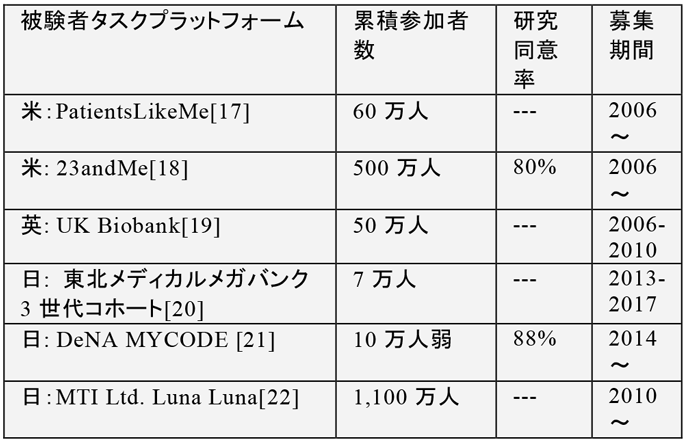
クラウドソーシングタスクで,募集期間での参加者数やデータ数を見積もることができればプロジェクトを企画しやすい.ライフサイエンス分野での代表的なクラウドソーシング研究である,生物生態調査研究とヒト被験者研究の事例を,それぞれ表2と表3にまとめた.
生物生態調査研究では,生物の目撃情報をクラウドワーカが収集して,タスクプランナーが対象生物の生息領域調査に用いる.対象生物目撃情報の注釈付けがクラウドワーカの主作業なので,生物生態調査研究のタスクはデータライフサイクルの中でアノテーションに分類できる.画像撮影を含むタスクの場合は,試料収集作業を含むのでサンプリングとアノテーションの両方に分類される.動機による分類は,無償と有償でそれぞれVoluntaryとIncentive by Moneyになる.プロジェクトの募集期間は,1日のみのイベント,数カ月のキャンペーン,期間オープン,とさまざまである.1日イベントでは,コアラ生態調査を目的とした南オーストラリア大学の観察イベントGreat Koala Count[16]が約千件のデータを収集している.eBirdの1日イベントGlobal Big Dayは,世界151カ国の参加者が約6千種の鳥を観察して,約5万件のデータが登録された.
一方,ヒト被験者タスクでは,クラウドワーカは生体試料提供や情報注釈を行う.生体試料提供のみのタスクは,データライフサイクルでサンプリングに分類される.試料提供とともに健康等情報をアンケート検査形式で提供する場合は,クラウドワーカが注釈付け作業も行うので,サンプリングとアノテーションの両方に分類される.動機による分類は,生物生態調査研究と同じくVoluntary(無償)とIncentive by Money(有償)になる.表3ではヒト被験者タスクを扱うプラットフォームをまとめた.募集継続中タスクの参加者数は,2018年4月時点の累積値を採用した.UK Biobankと東北メディカルメガバンクは国家プロジェクトで,他は企業のプロジェクトである.各プロジェクトは被験者であるクラウドワーカの仲介管理の役割を持ち,登録者に一時的な研究プロジェクトへの参加を募る.一時参加率の数値は参考になるので,個人健康情報共有プラットフォームPatientsLikeMeの報告論文[17]の値を紹介する.2016〜2017年の約14カ月で37万人に招待状を送付,2.6万人が参加を表明した.アンケート調査結果の提出人数は7,400人(完了率は82%)だった.37万人の登録数でも,クラウドワーカの6%しか招待状に応答しない.タスク完遂まで通しで計算した一時参加率は2%である.対象疾患の希少性や,所属するクラウドワーカの属性で参加率は変化すると考えられる.PatientsLikeMeの一時参加率の低さは,被験者を集めるコストが高いことを意味している.日本では,(株)MTIが提供する生理周期管理アプリLuna Lunaの登録者に遺伝子解析研究の参加を募ったところ,2週間で1万人以上の参加表明があったとの報告がある.
3.実践事例①光合成細菌の遺伝子機能注釈と参加特徴分析
第3章と第4章では,筆者らのクラウドソーシング研究結果を事例として紹介する.ここでは,光合成細菌の遺伝子機能注釈研究,クラウドワーカの貢献量の偏りとタスク配分の課題を中心に説明する.遺伝子機能注釈タスクは,データライフサイクルの分類では,アノテーションになる.また動機による分類ではVoluntary(無償)とIncentive by Money(有償)になる.
3.1 PCC 6803 株の遺伝子機能注釈クラウドソーシング
シアノバクテリアSynechocystis sp. PCC 6803は世界で初めてゲノム解析がされた光合成細菌であり,千葉県が母体のかずさDNA研究所が1996年に全塩基配列を発表した[23].PCC 6803株の約360万塩基対の配列から,3,725個の遺伝子領域が同定されている.我々は2015〜2016年に,PCC 6803株の遺伝子機能注釈の見直しを行った.具体的には,オンライン注釈システムを構築して,コミュニティ参加型クラウドソーシングにより1,096遺伝子のテキスト注釈を行った[24].再注釈の結果,機能未知の遺伝子は46.3%まで削減できた. 再注釈の参加者は,シアノバクテリアの研究コミュニティから募集された.コミュニティ参加型クラウドソーシングとは,ある専門知識を持つグループ(コミュニティと呼ぶ)内でのみ,参加者を募集する形式をいう.個々の参加者は微生物の異なる専門知識を持ち,自分の専門領域の最新知見は有するが,遺伝子の機能注釈作業の知識は持ち合わせていない.
3.2 遺伝子機能注釈の作業手順
再注釈では,まず遺伝子機能のテキスト注釈タスクをマイクロタスクに分割した.第1段階のアノテーションで,参加者はできるだけ専門領域や関連領域の遺伝子を担当した.第2段階でのアノテーションでは,遺伝子注釈に詳しい専門家が,参加者たちが専門外で付与した遺伝子機能の注釈内容を掘り下げて,品質向上を図った.
オンライン注釈システムが存在しなかった時代は,重要なモデル生物の遺伝子注釈作業は,ジャンボリと呼ばれる集会を開いて行った.ジャンボリでは,数日間30人ほどの参加者を宿泊施設にカンヅメにして,遺伝子機能注釈情報を編集する作業を分担する.しかし,注釈タスクの理解と合意形成の議論に時間がかかり,期間内に全遺伝子の注釈作業を終了することは困難だった.
3.3 PCC6803株遺伝子機能注釈の参加貢献量の偏り
オンライン注釈システムでは,参加者の作業履歴から注釈数(すなわち参加貢献量)をモニタリングする.多くの参加型プロジェクトの貢献量には,クラウドワーカ間で偏りが生じることが知られている[25].参加貢献の不均衡定量指標の1つに,Gini係数がある.Gini係数は,参加者間の貢献格差が大きいほど1に近付き,格差がないと0になる.オンライン注釈タスクでのGini係数の参考値は,市民科学ポータルサイトZooniverse掲載の7件のプロジェクトが0.77~0.91[26],Wikipedia で0.92以上[25]である.
参加者全員が同数の注釈マイクロタスクを完遂すればGini係数は0になる.PCC 6803注釈タスクでは,13人の参加者にタスクを均等配分しないで,自由投稿形式を採用した.58日間で1,149回投稿されたPCC 6803株注釈タスクのGini 係数は0.58だった[27].図1に他3件の投稿タスク(M1~M3)とともに,PCC6803タスク(A1)のクラウドワーカごとの貢献量とGini係数計算用のローレンツ曲線を示す.参加者の貢献数には偏りが見られ,一番投稿数の多いクラウドワーカは注釈全体の27%に貢献していた.
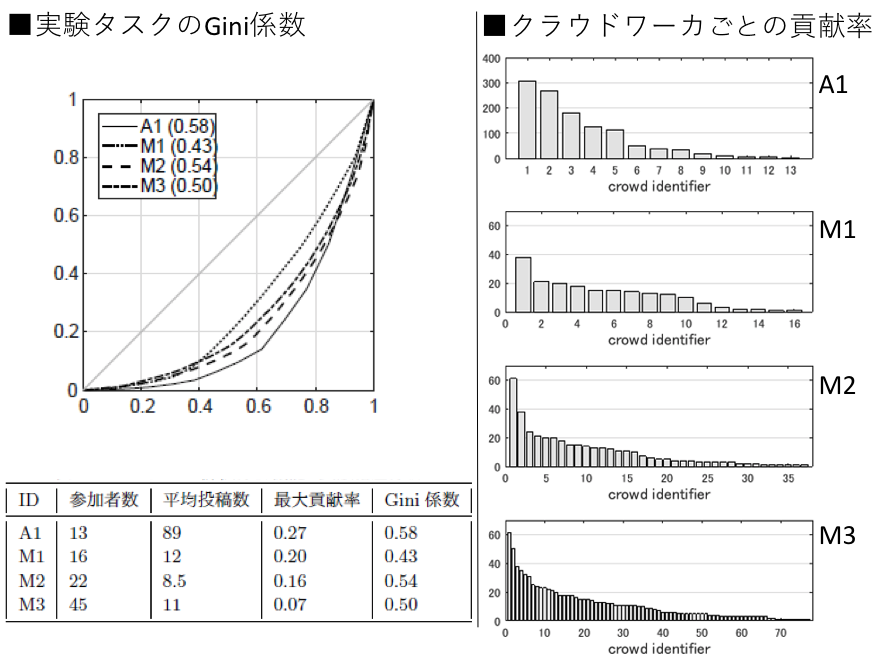
上記の参加貢献量分析の結果は,遺伝子機能注釈タスクにおいて,参加者へのタスクの均等配分が機能しない可能性を示唆している.昔のジャンボリ形式では,タスク割当は均等配分にしていた.注釈作業の早期終了の鍵は,貢献度が高い参加者への,より多くの活動機会の提供かもしれない.
3.4 コミュニティ参加型の国際塩基配列データベースの注釈品質事例
PCC 6803系統に限らず,遺伝子機能情報などの塩基配列注釈は,研究者が,論文公開用に国際塩基配列データベース(INSDC)へ登録する.INSDCは,研究者らのコミュニティ参加型で塩基配列の注釈付けが行われる.運営は,日本DNAデータバンク(DDBJ)[28]が欧州機関EBI・米国機関NCBIと分担している.DDBJに投稿された塩基配列注釈データは,専門キュレータによる注釈仕様の検証確認を経た後で,欧州・米国の投稿分と統合されてインターネット上で公開される.
塩基配列注釈データベースは,機械学習モデル訓練用の素材になる[29].INSDCの注釈情報は,入力任意条件の注釈属性に欠損が多い.また入力必須条件の注釈項目でも,表記揺れがあり,打ち間違い(Typo)も散見される.これら表記エラー以外に,登録内容自体(塩基配列や生物名)の間違いも存在する(これはINSDCだけでなく,参加型の生物試料分譲でも聞く話である.海外機関から寄託試料の植物種子を分譲してもらいDNA解析をしたところ,登録とは別品種と判明した等).
INSDCの登録時に,仕様検証チェックに引っかからなければ,注釈間違いはそのままインターネット上に公開される.コミュニティ参加型の遺伝子オントロジーデータベースの注釈エラー率は,13%~18%との報告がある[30].我々の調査でも,生物試料のINSDCデータベースBioSampleから,微生物の温度の注釈属性9,269件を抽出したところ,単位情報の表記揺れを含めると全体の20%が注釈エラーを含んでいた.注釈のデータクレンジングは,修正ツール(OpenRefine等)を使用している.
3.5 非専門家クラウドワーカによるライフサイエンス専門用語の注釈品質
PCC 6803株遺伝子機能注釈の参加者は,研究コミュニティの所属者で,ライフサイエンスの背景知識を持つ.また注釈品質は2段階アノテーションの仕組みで担保していた.2段階アノテーションの仕組み(1段階目で投稿された低品質注釈を,2段階目に専門家が検証して注釈品質を向上させる)は,国際塩基配列データベースとeBirdの両方に採用されている.ここで1つの注釈データの構築に,2名が関係していることに注意する.
ライフサイエンスの背景知識を持たない非専門家のクラウドソーシングの場合は,生成された複数人分の注釈情報を集約(Aggregate)することで注釈品質を向上させる.GoodらのAMTを用いたNCBI Disesase Ontologyコーパスを用いた疾患名注釈の研究[12]では,145名のクラウドワーカを雇用して平均注釈精度(F値)が0.76で,6人分の多数決方式(Simple Majority Voting)で0.87まで注釈精度が向上している.
筆者らは,京大鹿島研との共同研究で,日本の商用クラウドソーシングプラットフォームを活用して,非専門家の注釈品質評価を行った[31].タスクは,遺伝子機能注釈ではなく,GENIAコーパス(http://www.geniaproject.org/)を使い分子生物学用語の抽出とした.28名の非専門家クラウドワーカと3名の専門キュレータの平均注釈精度(F値)は,0.30と0.46だった.クラウドワーカにTOEIC得点と生命科学の学歴(大学以上)のアンケートを取ったが,注釈精度との相関は見られなかった.作業時間のみ,注釈精度と中程度の正の相関が見られた(ピアソンの積率相関係数r=0.49, P値=0.009).28名の中で上位成績のクラウドワーカ2名(F値=0.50, 0.49)は,専門キュレータ上位者2名の成績(F値=0.54, 0.52)に近い性能だった.しかし,クラウドワーカと専門キュレータの平均注釈精度は0.16も乖離している.Goodらの研究では,最初に346人に試験を課して,合格した145人(合格率42%)のみが注釈作業を実施していた.非専門家へのライフサイエンス分野の専門的注釈作業の委託については,このような作業能力の事前スクリーニングが課題となるだろう.
4.実践事例②植物DNA配列注釈の機械学習コンペティション
ライフサイエンス研究分野では,実験計測機器の技術革新により,実験研究者が大量のデジタルデータを容易に得られる時代になっている.計測データの機械学習モデル化に興味がある実験研究者は多いが,機械学習のスキルを持つ共同研究相手を探すことは容易ではない.共同研究者探しの解決方法として,群衆へ課題をアウトソーシングするクラウドソーシングの活用がある.筆者らは,DNA配列注釈の予測モデルの精度を群衆が競う機械学習コンペティション(名称: DDBJデータ解析チャレンジ)を企画して,2016年夏に実施した[32].本タスクは,データライフサイクルではモデリングに分類され,動機では競争に分類される.
4.1 DDBJデータ解析チャレンジの開催
DDBJデータ解析チャレンジの課題は「シロイヌナズナDNA配列からのクロマチン特徴注釈の予測」である.DDBJ保有配列データの植物ゲノムの断片配列に,特定のクロマチン特徴注釈領域が含まれるか否かを予測する.クロマチンとはDNAとタンパク質の複合体で,遺伝子の発現の制御機能を持つ.クロマチン関連因子が存在するゲノム領域の特定は,遺伝子発現制御の理解に繋がる.訓練データは,クロマチン特徴注釈データベースChIP-Atlas[33]の未掲載生物種を対象に,機械学習コンペティション用にオリジナルで作成した.ChIP-Atlasは,DDBJ公開の次世代DNAシークエンサ配列アーカイブSequence Read Archive (SRA) [28]を解析して構築された,2次注釈データベースである.
クラウドワーカが構築した予測モデルの投稿管理とリーダーボード表示(モデル精度視覚化)は,京大鹿島研の教育用機械学習コンペティション基盤であるビッグデータ大学[34]で行った.さらにDDBJ解析チャレンジの参加者には,計算機資源も提供した.DDBJで管理している遺伝研スーパーコンピュータ[35]のGPU計算ノード16台を,チャレンジ参加者であるクラウドワーカへ期間中のみ無償で提供した. またR, Python, Caffe, Chainerのソフトウェアをチャレンジ開催に合わせて遺伝研スパコンに整備した.MathWorks Japan社とは開催期間中に,遺伝研スパコンGPU環境と,ローカルPC環境の両方で使えるMATLABライセンスを無償提供してもらうスポンサーシップ契約を結んだ.
4.2 参加者数と上位入賞モデルの予測精度
機械学習コンペティションを57日間実施した結果,クラウドワーカ総数は38名でモデル投稿回数は延べ360回だった.DDBJデータ解析チャレンジへの参加の呼びかけは,複数のバイオインフォマティクス分野のメーリングリストと,参加基盤のビッグデータ大学内で告知を行っていた.上位3位までの入賞者全員が,生命情報科学の背景知識を持っていた.4位の参加者は情報科学の専門だった.5位で学生1位の参加者は,生命情報科学の専門だった.
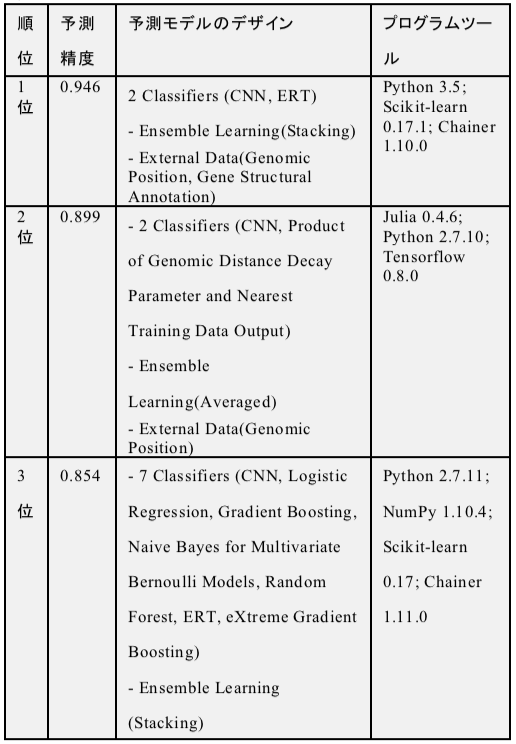
優勝モデルの予測精度(評価尺度はArea Under the Curve, AUC)は0.95である.表4に上位3モデルの予測精度・モデルデザイン・プログラムツールをまとめた.最初の投稿モデルの予測精度は0.65だったので,コンペティション期間で0.30予測性能が向上したことになる.優勝モデルは 畳み込みニューラルネットワークを含む2種類の分類器をアンサンブル学習で組み合わせており,特徴として外部データ(ゲノム位置情報と遺伝子構造注釈情報)を採り入れていた.2位の予測モデルも,ゲノム位置情報の外部データを採用していた.
参加貢献の定量分析として,参加者の投稿回数と投稿モデル精度の間でピアソン相関係数を計算したところ,弱い正の相関r=0.35 (P値=0.03) が見られた.文献では,Kaggle (商用機械学習コンペティション基盤, https://www.kaggle.com/) の競技結果を分析して投稿回数と予測モデル精度に,正の相関が見られたという報告もあるが,負の相関の報告も存在する.今後データが蓄積していくことで,参加者の投稿回数と予測モデル精度の関係が明らかになっていくだろう.
4.3 機械学習コンペティション開催の知見と課題
ここでは,ライフサイエンス分野の機械学習コンペティションの開催に興味がある読者に向けて,訓練データの準備コスト,不正防止用情報マスキング,競技用ルール設定,訓練データ量の削減の4つの知見を紹介する.
① 訓練データの準備コスト
訓練データの準備に時間がかかった.訓練データには未公開データを自作で準備した.チート行為の対策として,参加者が競技タスクの正答を簡単に探せないように,訓練データを準備する必要がある.一番簡単な準備方法は,実験研究者から未公開データを受け取り,訓練データにすることである.しかし,複数名の実験研究者に問い合わせたが,実験データの公開許可は得られなかった.ChIP-Atlas作者の沖博士は機械学習コンペティションに理解があり,未解析生物種の自前データ作成を条件に利用許諾が得られた.このように,実験研究者からの未公開データ提供は容易でなく,タスクの準備には時間がかかるだろう.
② 不正防止用情報マスキング
訓練データは,不正防止用に情報マスキングを行った.具体的には,ChIP-Atlasの元データベースであるSRAの検索画面で配列を特定できないように,訓練データの実験条件(Antigen x Cell Type)を非公開とした.さらにタスクの説明事項に,SRAデータの参照配列アライメント解析によりテストデータの正答を算出する行為の禁止を入れた.
③ 競技用ルール設定
外部データや転移学習用訓練済モデルの利用可否など,競技ルールを細かく設定する必要がある.コンペティション開始時にこれらのルールを明記していなかったので,参加者から質問がきた.逐次ルールを決めて,質問への返信の形でFAQに掲載した.ルールの条件決めにはKaggleの競技ルール規定を参考にした.
④訓練データ量の削減
計算機資源に依存して,訓練データ量を削減した.参加者に提供した遺伝研スパコンの計算ノード数は限られていたために,訓練データのサイズは,手元のノートPCで計算が可能なサイズに絞った.訓練データ量の増加は,計算時間増大につながり,参加者間で計算機資源の取り合いになる.GPU計算ノードは,遺伝研スパコンの研究利用向け計算ノードの一部を切り出しており,簡単に割当数を増やせない.訓練データの規模を増やすことは,今後の課題である.
5.研究倫理審査承認とクラウドソーシング研究
ライフサイエンス研究分野では,クラウドワーカの利用は研究倫理審査の承認が必要と考えた方がよい.ライフサイエンス分野の多くの研究雑誌が,投稿規定にクラウドワーカ参加研究の研究倫理審査承認を課しているからである.ある雑誌では,投稿規定に次のように記載されている.「Human Participants, Human Data or Human Tissueを含む研究は,ヘルシンキ宣言[36]にしたがって実施されていることを,研究倫理審査委員会から承認を得る必要がある」.ヘルシンキ宣言とは,World Medical Associationが提供するヒトを対象とする医学研究の倫理指針である.以下に研究倫理審査委員会について説明する.
5.1 人を対象とする研究倫理審査委員会
DDBJ Challengeはクラウドソーシング研究で,参加者であるクラウドワーカは,機械学習モデルの構築作業を行った.クラウドワーカは被験者(Human Subjects)ではないが,投稿規定での参加者(Human Participants)に該当する.Human Participantsを扱うために,研究開始前に国立遺伝学研究所の「人を対象とする研究倫理審査委員会」より研究実施の承認(番号No.28-3)を得た.もし唾液等のヒト試料を使いヒトゲノム遺伝子解析研究も同時に行う場合には,クラウドソーシング研究は上記委員会ではなく,より厳格な「ヒトゲノム・遺伝子解析研究倫理委員会」で審査される.研究倫理審査委員会は,英語ではInstitutional Review Board(IRB)と表記される.研究雑誌側は,IRBの承認番号と,研究同意書 (Informed Consent:IC)情報の2項目を,論文原稿に記載することを筆者に求めてくる.ICについて次に説明する.
5.2 インフォームド・コンセント
研究倫理審査委員会では,クラウドワーカへ提示する研究同意書のチェックが行われる.次に,国立遺伝学研究所IRBで承認されたIC の項目を示す.
- 1) 研究目的・協力方法・実施体制・研究期間について
- 2) 研究が国立遺伝学研究所の倫理審査委員会で承認された上で,開始されること
- 3) 研究成果の公表について
- 4) 利益・不利益について
- 5) 研究データの個人情報保護および匿名化について
- 6) 研究のデータの保管と廃棄について
インターネット上で参加者を募る場合には,紙で研究同意書に署名してもらうことができない.代わりに,ICはオンラインWebサイト上に掲載する.Webサイト上に同意ボタンを提示する方式等を使うことで,ICへの同意と見なす.5)の個人情報保護と6)のデータ保管・廃棄については,具体的な情報管理方法をICで説明する必要がある.注意点として,研究途中で参加者が離脱可能な仕組みを提供する必要がある.つまり,研究同意後に参加を辞退するオプトアウトの手続き方法を提示しておく必要がある.
5.3 グレーゾーンのクラウドソーシング研究のIRB申請
研究内容が医学に関係がなく,社会心理学研究のような被験者扱いでもない場合のクラウドソーシング研究は,IRB承認が必要だろうか.特にクラウドワーカが注釈作業やモデリング作業で参加する場合には,IRB申請が必要かは,グレーゾーン扱いで判断が難しい.このようなグレーゾーン研究も,最近はIRB承認が推奨されている[37].もし読者がグレーゾーンのクラウドソーシング研究を計画して最終的にライフサイエンス系研究雑誌に投稿予定なら,リスク回避のためにIRB承認取得をお勧めする.筆者らはライフサイエンス系の海外雑誌にグレーゾーン研究を投稿したが,投稿窓口で差し戻された.窓口担当者と何度も交渉したが,IRB承認番号の要求一辺倒で,結局,原稿の受け付けさえしてもらえなかった.
5.4 IRB承認は時間を遡れない
研究論文誌の投稿規定には,研究倫理委員会の「遡及的な承認」を認めない,と明記している場合が多い.通常のIRB審査は,時間を遡った承認効果を認めていない.これより研究実施者は,クラウドソーシング研究の開始前に,IRB申請を済ませて研究倫理の承認を得る必要がある.もしIRBが実施者の所属組織に存在しない場合には,研究倫理審査委員会を新規に創設してもらうか,IRBが存在する別機関の共同研究者を探して申請する必要があるだろう.
6.おわりに
ライフサイエンス研究分野における,クラウドソーシング・タスクの分類について紹介するとともに,募集期間と参加者数等の参考情報を紹介した.またクラウドソーシング研究の実践事例として,遺伝子機能アノテーションとDNA配列注釈モデリングを紹介し,クラウドワーカ活用で得られた知見をまとめた.さらに,ライフサイエンス分野でクラウドワーカ活用研究を研究雑誌に投稿する場合には,研究倫理審査の承認番号が要求されることを説明した.本稿の解説により研究倫理審査に対する理解が深まり,ライフサイエンス分野のクラウドソーシング研究が増えることを期待している.
謝辞 クラウドワーカとして研究に参加していただいた皆様,東京医科歯科大学の田中博特任教授,国立遺伝学研究所の高木利久教授,有田正規教授,大久保公策教授,小笠原理特任准教授,京都大学の鹿島久嗣教授,産総研人工知能研究センター瀬々潤研究チーム長,筑波大学の馬場雪乃准教授,九州大学の沖真弥助教,日本DNAデータバンクの皆様,MathWorks Japan社の大谷卓也様,天野光様に感謝いたします.
参考文献
- 1)Howe, J. : Crowdsourcing : Why the Power of the Crowd is Driving the Future of Business, Crown Publishing Group New York (2008).
- 2)Rees, M. A. : Longitude Prize for the Twenty-First Century, Nature, 509, 401 (2014).
- 3)宮崎佑介:市民科学と生物多様性情報データベースのかかわり, 日本生態学会誌, Vol.66, pp.237-246 (2016).
- 4)Sullivan, B. L. et al. : eBird : A Citizen-based Bird Observation Network in the Biological Sciences, Biological Conservation, 142, 2282 (2011).
- 5)Laaksonen, M. et al. : Crowdsourcing-based Nationwide Tick Collection Reveals the Distribution of Ixodes Ricinus and I. Persulcatus and Associated Pathogens in Finland, Emerg Microbes Infect 10:e31 (2017).
- 6)Yank, V. et al. : Crowdsourced Health Data : Comparability to a US National Survey, 2013-2015, Am J Public Health, 107,1283 (2017).
- 7)Quade, P. et al. : A Platform for Crowdsourced Foodborne Illness Surveillance: Description of Users and Reports, JMIR Public Health and Surveillance, 5:e42 (2017).
- 8)Berman, F. et al. : Realizing the Potential of Data Science, Communications of the ACM, 61:67-72 (2018).
- 9)Good, BM. and Su, A. : Crowdsourcing for Bioinformatics, Bioinformatics, 29:1925 (2013).
- 10)Hoffmann, R. : A Wiki for the Life Sciences where Authorship Matters, Nature Genetics, 40, 1047 (2008).
- 11)Prill, RJ, et al. : Crowdsourcing Network Inference : the DREAM Predictive Signaling Network Challenge, Sci Signal, 4, mr7 (2011).
- 12)Good, BM, et al. : Microtask Crowdsourcing for Disease Mention Annotation in Pubmed abstracts, Pac Symp Biocomput, 282 (2015).
- 13)Cooper S, et al. : Predicting Protein Structures with a Multiplayer Online Game, Nature, 466, 756 (2010).
- 14)Lee J, et al. : RNA Design Rules from a Massive Open Laboratory, Proc Natl Acad Sci U S A., 111, 2122 (2014).
- 15)Tackett, S. et al. : The reCAPTCHA of Medical Education, Med Teach 1460463 (2018).
- 16)Sequeira, AM, et al. : Distribution Models for Koalas in South Australia Using Citizen Science Collected Data, Ecol Evol, 4, 2103 (2014).
- 17)Wicks P, et al. : Scaling PatientsLikeMe via a “Generalized Platform” for Members with Chronic Illness : Web-Based Survey Study of Benefits Arising. J of Med Internet Res, 2, e175 (2018).
- 18)23andMe : https://mediacenter.23andme.com/company/about-us/
- 19)Sudlow, C. et al. : UK Biobank : An Open Access Resource for Identifying the Causes of a Wide Range of Complex Diseases of Middle and Old Age, PLoS Med, 12:e1001779 (2015).
- 20)Kuriyama, S. et al. : The Tohoku Medical Megabank Project : Design and Mission, J Epidemiol, 26:493 (2016).
- 21)DeNA MYCODE : https://mycode.jp/
- 22)Khor, S-S. et al. : Genome-wide Association Study of Self-reported Food Reactions in Japanese Identifies Shrimp and Peach Specific Loci in the HLA-DR/DQ Gene Region, Sci Rep, 8:1069 (2018).
- 23)Kaneko, T. et al. : Sequence Analysis of the Genome of the Unicellular Cyanobacterium Synechocystis Sp. Strain PCC6803, DNA Res, 3, pp,109-136 (1996).
- 24)Fujisawa, T. et al. : CyanoBase : A Large Scale Update on Its 20th Anniversary, Nucleic Acids Res, 45, D551-554 (2017).
- 25)Ortega, F. et al. : On the Inequality of Contributions to Wikipedia. Hawaii Int. Conf. on System Sciences, 304 (2008).
- 26)Sauermanna, H. et al. : Crowd Science User Contribution Patterns and Their Implications, PNAS, 112 pp.679-684 (2015).
- 27)神沼英里 他:CrowdR&D:クラウド協働評価のための参加型R&Dプロジェクト情報統合基盤,第30回日本人工知能学会全国大会,1L4-OS-09a-6 (2016).
- 28)Kodama, Y. et al. : DNA Data Bank of Japan : 30th anniversary, Nucleic Acids Res, 46:D30-35 (2018).
- 29)神沼英里 他:ディープラーニングを用いたDNA配列からの微生物生態属性値の予測,第12回日本ゲノム微生物学会年会, 2St1-07 (2018).
- 30)Jones, CE. et al. : Estimating the Annotation Error Rate of Curated GO Database Sequence Annotations, 8, 170, BMC Bioinformatics (2007).
- 31)神沼英里 他:遺伝子構造キュレーションのクラウドソーシング・タスク設計,第28回日本人工知能学会全国大会,1J5-OS-18b-3 (2014).
- 32)DDBJ Data Analysis Challenge : https://www.ddbj.nig.ac.jp/activities/ddbj-challenge.html
- 33)Oki, S. et al. : Integrative Analysis of Transcription Factor Occupancy at Enhancers and Disease Risk Loci in Noncoding Genomic Regions, bioRxiv doi:10.1101/262899 (2018).
- 34)Baba, Y. et al. : Data Analysis Competition Platform for Educational Purposes : Lessons Learned and Future Challenges, In Proceedings of the 8th Symposium on Educational Advances in Artificial Intelligence (EAAI) (2018).
- 35)Ogasawara, O. et al. : DDBJ New System and Service Refactoring, Nucleic Acids Res, 41, D25-29 (2013).
- 36)Hellmann, F. et al. : 50th Anniversary of the Declaration of Helsinki, Archives of Med Res 45, pp.600-601 (2014).
- 37)Graber, M. A. and Graber, A. : Intenet-based Croudsourcing and Research Ethics : The Case for IRB Review, J Med Ethics, 30, pp.115-118 (2013).
東京医科歯科大学 医療データ科学推進室 特任講師.医療・創薬データサイエンスコンソーシアム担当教員. 国立遺伝学研究所 生命情報研究センター 大量遺伝情報研究室 外来研究員と,産業総合研究所 人工知能研究センター 機械学習チームの協力研究員を兼務.生命情報データの自動注釈解析を専門に,データサイエンス教育やDNAデータバンク事業に従事.
藤澤 貴智(非会員)tf@nig.ac.jp国立遺伝学研究所 生命情報研究センター 大量遺伝情報研究室 特任研究員.ライフサイエンス統合推進事業において,微生物のリファレンスとして重要な菌株あるいは現象についての情報の高度化およびゲノム・メタゲノム情報を基盤としたデータサイエンスを加速させる微生物統合データベースの高度実用化開発に従事.
中村 保一(非会員)yn@nig.ac.jp国立遺伝学研究所 生命情報研究センター 大量遺伝情報研究室 教授(兼 総合研究大学院大学 生命科学研究科 遺伝学専攻 教授).DDBJセンター データベース部門長.ゲノム情報解析と配列データベースの作成を専門とする.
編集担当:濱崎 雅弘(産業技術総合研究所)